Evoluzione umana: l’importanza di analizzarne le basi molecolari Understand article
Tradotto da Sara Cogliati. Nel secondo articolo, Jarek Bryk ci spiega come gli scienziati studino a fondo i nostri geni per capire le basi molecolari di un particolare adattamento evoluzionistico del genere umano.

concessa da Henrik500
/ iStockphoto
Il DNA di tutti gli organismi conserva informazioni sulla loro storia evolutiva sia recente che passata. Studiando le caratteristiche comuni e le variazioni delle varie sequenze, comparandole tra diversi individui e specie, è possibile scoprire cosa è accaduto ai diversi organismi durante l’evoluzione. In questo modo è possibile individuare quali geni o quali frammenti del genoma hanno probabilmente rappresentato per quegli individui o specie che li possedevano un vantaggio evolutivo in termini di sopravvivenza ed efficienza riproduttiva (vedi il glossario per i termini in grassetto).
In un precedente articolo (Bryk, 2010), ho descritto alcuni esempi di variazione genica vantaggiosi per l’uomo e per altri organismi. Dimostrare quali cambiamenti genici possono avere degli effetti positivi è molto complicato, soprattutto nell’uomo, ma ancora più difficile è dimostrare il meccanismo mediante il quale questi cambiamenti possono migliorare la sopravvivenza e la riproduzione dei vari organismi.
In questo articolo, descriverò uno dei tanti approcci che può essere usato dagli scienziati prima, per identificare regioni del nostro genoma che potrebbero averci aiutati nella sopravvivenza e nella riproduzione e successivamente, per dimostrare come queste regioni genomiche possano aver avvantaggiato i nostri antenati.
Il confronto fra sequenze di DNA di molti individui appartenenti a differenti popolazioni, è uno dei modi per identificare regioni genomiche potenzialmente vantaggiose. Per esempio, in uno scenario molto semplificato, se una popolazione subisce una specifica pressione selettiva (per esempio, l’elevata radiazione UV in un’area molto soleggiata) la sequenza di DNA responsabile di quell’ adattamento specifico (per esempio, il colore della pelle più scuro), sarà diversa rispetto a quella che invece è presente in un’altra popolazione che non è oggetto di quella particolare pressione selettiva.
Adattato con il permesso di Macmillan Publishers Ltd: Nature, Sabeti et al. (2007), © 2007
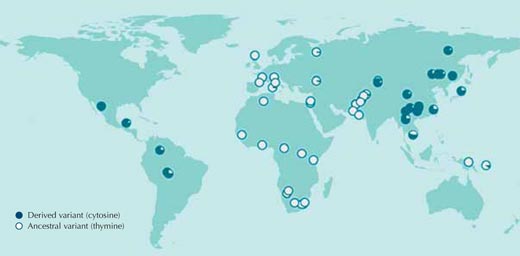
Nella maggior parte dei casi, purtroppo, noi non conosciamo quale sia la pressione selettiva a cui le popolazioni sono state sottoposte e neppure quale siano i geni responsabili dell’adattamento. Ciò nonostante, in ogni caso, si incomincia confrontando le sequenze di DNA tra le varie popolazioni senza ipotizzare nulla su ciò che potremmo trovare. La figura 1 mostra una di queste analisi per un singolo nucleotide del genoma umano.
La situazione in cui in una particolare posizione della sequenza di DNA si trovano nucleotidi diversi tra i vari individui, è definita polimorfismo a singolo nucleotide(SNIP, pronuncia ‘snip’); tre milioni di questo tipo di varianti del genoma umano sono catalogate nell’archivio pubblico HapMapw1. Lo SNP illustrato in Figura 1, rs3827760, è stato trovato in due varianti o alleli: in quel punto della sequenza, è possibile trovare una di queste due basi: una timidina (T) o citosina (C).
Ogni cerchio in figura rappresenta una singola popolazione e descrive la frequenza dei due possibili alleli in quella popolazione.
L’allele contenente la timidina è presente in tutti i campioni africani e nella maggior parte di quelli europei mentre è completamente assente negli asiatici dell’Est e tra gli americani nei quali è prevalente la citosina (Sabeti et al., 2007,2006; Xue et al., 2009).
Se si facesse un confronto fra tutti gli altri tre milioni di SNP presenti su HapMap, si potrebbe osservare che la distribuzione delle varianti rs3827760 tra le popolazioni umane è molto inusuale. Per questo motivo lo SNP rs3827760 merita un’osservazione più dettagliata, anche se la distribuzione tra le popolazioni non ci suggerisce nulla riguardo ai potenziali vantaggi evolutivi delle due varianti (il loro valore adattativo), o se esse sono realmente adattative. Tutto quello che sappiamo, per ora, è che per qualche ragione, la timidina che originariamente era presente in questa posizione in una popolazione ancestrale dell’Africa mutò in citosina e questo cambiamento si diffuse negli asiatici dell’est e negli americani. Persino le stime riguardo a quando questo cambiamento è avvenuto sono molto imprecise: più o meno tra 1000 e 70000 anni fa, tutti gli individui delle popolazioni nell’Asia dell’Est hanno acquisito la variante con citosina.
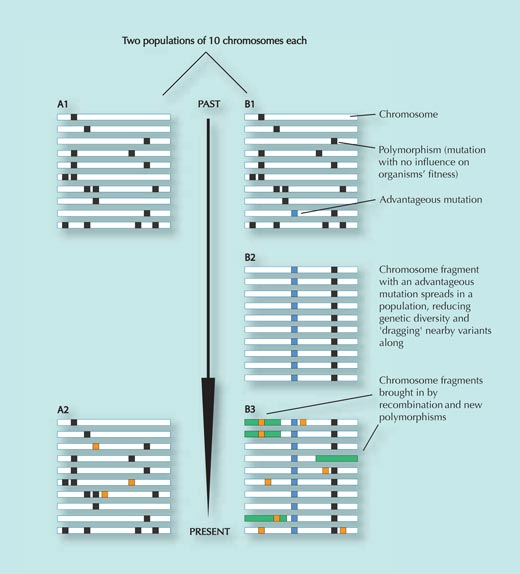
La popolazione A non è stata oggetto di nessuna pressione selettiva ed è rimasta relativamente invariata lungo il tempo, a parte l’acquisizione di alcuni cambiamenti genetici casuali che non hanno influenzato la fitness (quadratini arancioni nel pannello A2; le variant che riducono la fitness sono eliminate dalla popolazione)- paragona pannello A1 e A2.
La popolazione B si sposta in un ambiente diverso dove incontra una nuova pressione selettiva. In questo nuovo ambiente, una particolare variazione genica (il quadrato blu nel pannello B1) rappresenta un vantaggio per gli individui che la possiedono e così quella variazione può diffondere velocemente nella popolazione (gli individui che la possiedono hanno più progenie). Le varianti geniche che si trovano vicine a questo SNP vengono trasmesse insieme ad essa (più le due varianti si trovano vicine, minore sarà la possibilità che queste vengano separate durante lo scambio tra cromosomi materni e paterni – vedi Figura 3).
Il risultato di questa rapida trasmissione di sequenze di DNA nella popolazione è una riduzione della diversità genetica in quella regione cromosomica; la maggior parte degli individui potrà avere lo SNP vantaggioso insieme alle varianti geniche vicine ad esso (compara il pannello B1 e B2). Questo processo si svolge molto velocemente.
Dopo qualche tempo, comunque, le nuove mutazioni e i nuovi eventi di ricombinazione introducono nuove varianti (rettangoli verdi e quadrati arancioni nel pannello B3). Quanto più la diffusione della variazione originariamente selezionata è remota nel tempo, tanto più difficile sarà individuarla poiché la ridotta variazione genica (B2) sarà probabilmente mascherata (B3)
Immagine gentilmente concessa da Jarek Bryk
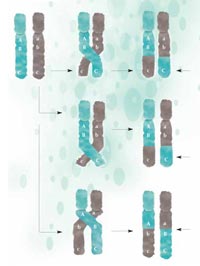
ricombinazione, parti di DNA
sono scambiati tra i
cromosomi materni (verde) e
paterni (grigio)e passano alla
nuova generazione in una
nuova configurazione. Più le
due regioni sono vicine,
meno andranno incontro a
separazione durante la
ricombinazione: A e B sono
più vicini di B e C, così più
difficilmente saranno
separate. Cliccare
sull’immagine per ingrandirla
Immagine gentilmente
concessa da Nicola Graf
Come possiamo capire se questo fenomeno è frutto di una selezione positiva (la citosina ha dato un vantaggio evolutivo nell’Asia dell’Est e in America) o è semplicemente frutto del caso? Per verificare se il cambiamento della base di DNA (timidina o citosina) è stato selezionato positivamente, abbiamo spostato la nostra attenzione alla sequenza attorno allo SNP. Qualora la sequenza attorno al rs3827760 fosse stata simile in tutte le popolazioni, non avremmo avuto nessuna conferma dell’effetto dello SNP sulla fitness dell’organismo. Se invece, una popolazione (quella dell’Asia dell’est, per esempio) fosse stata esposta ad una pressione selettiva e il rs3827760 avesse contribuito allo sviluppo di una adattamento per una pressione selettiva, le sequenze attorno allo SNP, sarebbero state differenti tra le popolazioni. Per capirne il perchè, vedi Figura 2.
Quando le sequenze di DNA attorno al rs3827760 furono comparate, divenne ovvio che la variazioni genica delle sequenze attorno alla variante con citosina nelle popolazioni dell’Asia dell’Est è infatti molto più bassa rispetta alla diversità attorno alla variante con timidina trovata nelle popolazioni africane ed europee (gli Americani non furono testati). Questo suggerisce che la selezione positiva fu responsabile della variante con citosina diffusa poi nelle popolazioni Est asiatiche. Ma questo SNP è stato effettivamente selezionato? Questo polimorfismo, in sostanza, ha un effetto biologico?
Non tutte le mutazioni nelle sequenze del DNA hanno un effetto sulla sequenza proteica: la maggior parte degli SNP catalogati nel database HapMap sono localizzati in sequenze non codificanti del genoma (e.g. tra due geni) oppure sono sinonimi e cioè sono localizzati in parti codificanti del genoma ma non causano un cambiamento della sequenza proteica (vedi Figura 4).
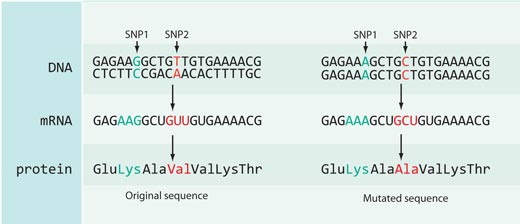
Immagine gentilmente concessa da Jarek Bryk
Nel caso del polimorfismo rs3827760, siamo stati fortunati perché è localizzato in una parte codificante del genoma- verso la fine di un gene chiamato EDAR, il quale è coinvolto nello sviluppo dei follicoli piliferi, delle ghiandole sudoripare e dei denti. Inoltre, il cambiamento da timina a citosina risulta in un cambiamento della sequenza proteica: gli Africani e gli Europei (che hanno la variante di SNP con timina) hanno l’aminoacido valina alla posizione 370 della proteina, mentre gli Asiatici e gli Americani (che hanno invece il nucleotide citosina) hanno l’aminoacido alanina. Questa parte della proteina è coinvolta nell’interazione con altre proteine e inoltre sono note mutazioni in questo dominio correlate a displasie ectodermiche caratterizzate da uno sviluppo anomalo di denti, capelli e ghiandole sudoripare, sia nell’uomo che nel topo (vedi Figura 5). Questo suggerisce che il cambiamento di un singolo aminoacido nella posizione 370 non è solo un cambiamento della sequenza proteica ma causa un cambiamento importante della funzione della proteina con un impatto sulle caratteristiche dell’organismo stesso.
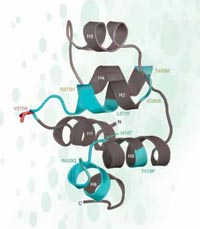
del dominio della proteina
EDAR. Le mutazioni
sottolineate in verde causano
nell’uomo la displasia
ectodermica. Il putativo SNP è
sottolineato in rosso
Adattamento permesso da
Macmillan Publishers Ltd:
Nature, Sabeti et al. (2007), ©
2007
Per verificare se il cambiamento nella sequenza proteica potesse veramente modificarne la funzione, abbiamo eseguito degli esperimenti mirati a studiare la via di segnale biochimico di cui la proteina EDAR fa parte: una serie di reazioni che sono coinvolte nello sviluppo dei follicoli piliferi, delle ghiandole sudoripare e dei denti. I risultati degli esperimenti hanno evidenziato che la variante proteica con alanina (trovata negli Asiatici dell’Est e negli Americani, codificata dalla variante SNP con citosina) rende la via di segnale più attiva rispetto alla variante con valina (trovata negli Africani e negli Europei, codificata dalla variante timina). Questo risultato ha un legame con la struttura del capello e dimostra che le persone con la variante alanina hanno un capello più spesso rispetto alle persone con la variante valina. Per una dimostrazione più diretta, sono stati geneticamente modificati dei topi in modo tale da rendere la via di segnale di EDAR più attiva. Questi topi hanno il manto visibilmente più folto con un pelo più spesso e ghiandole salivarie più grandi rispetto ai topi che possiedono un’attività di EDAR normale (Chunyan et al., 2008; Chang et al., 2009).
Queste osservazioni sperimentali suggeriscono che le due varianti SNP (che contengono o a timina o la citosina), hanno un effetto sia sulla struttura che sulla funzione della proteina EDAR. Queste modificazioni possono portare a differenze fisiche nell’uomo, essi infatti modificano lo spessore dei capelli e la dimensione delle ghiandole salivarie.
Le differenze nelle sequenze del DNA che noi ora osserviamo sono tracce storiche degli esperimenti della natura. Noi possiamo solo speculare sulla pressione selettiva a cui le popolazioni asiatiche e americane sono state sottoposte e che ha permesso la diffusione dell’allele con citosina. Grazie alla combinazione di studi genomici, ai risultati ottenuti in laboratorio e ai modelli animali creati, è stato possibile verificare alcune ipotesi sul ruolo funzionale delle differenze geniche tra le popolazioni o le specie. Usando questi approcci, ci è possibile capire le cause molecolari dei vari adattamenti naturali a cui i nostri antenati e gli altri organismi sono stati sottoposti. Ciò sottolinea così come noi ci adattiamo ai continui cambiamenti ambientali.
Glossario
Valore adattativo: una caratteristica ha un valore adattativo se conferisce ad un individuo la capacità di sopravvivere e di riprodursi meglio in un dato ambiente rispetto agli individui che non possiedono quella caratteristica. Più specificatamente, una caratteristica è adattativa se aumenta la fitness.
Fitness: è un termine specifico della biologia evoluzionistica e della genetica delle popolazioni difficile da definire; descrive la media numerica di nuovi nati di una generazione associati ad un fenotipo rispetto ad un altro genotipo presente nella popolazione totale. Il genotipo che produce più nuovi nati ha una maggiore finess.
Genoma: DNA totale di un organismo. Normalmente negli eucarioti lo si considera come il DNA nucleare, per distinguerlo dal DNA mitocondriale o dei plastidi. Per ulteriori informazioni consultare “What is a genome” (“che cosa è il genoma”) nel sito internet della Biblioteca Nazionale Medica Statunitensew2.
Selezione positiva: la selezione positiva è uno dei meccanismi dell’evoluzione. Descrive la diversa sopravvivenza e riproduzione degli individui in un determinato ambiente. La selezione naturale è definita positiva quando promuove certe caratteristiche che aiutano gli individui a sopravvivere e a riprodursi meglio che altri.
Pressione selettiva: caratteristica dell’ambiente (e.g. temperatura, presenza di parassiti, predazione o aggressioni da parte di membri della stessa specie) che determina una sopravvivenza e una capacità riproduttiva differente tra gli individui.
SNP: polimorfismo a singolo nucleotide ovvero singola base della sequenza del DNA che differisce tra gli individui. Si pronuncia ‘snip’.
References
- Bryk J (2010) Natural selection at the molecular level. Science in School 14: 58-62.
- Chang SH et al. (2009) Enhanced EDAR signalling has pleiotropic effects on craniofacial and cutaneous glands. PLoS ONE 4(10): e7591. doi:10.1371/journal.pone.0007591
- Questo articolo descrive il fenotipo di diverse ghiandole di topi con un aumento della via di segnale di EDAR e avanza ipotesi su quali caratteristiche potrebbero aver avuto una selezione positiva nella storia dell’umanità. L’articolo è disponibile gratuitamente nel sito internet del giornale: www.plosone.org
- Chunyan M et al. (2008) Enhanced ectodysplasin-A receptor (EDAR) signaling alters multiple fiber characteristics to produce the East Asian hair form. Human Mutation 29(12): 1405-1411. doi: 10.1002/humu.20795
- L’articolo riporta in dettaglio gli studi eseguiti in vitro con la protein EDAR e con i topi transgenici, con figure e foto molto interessanti.
- Pongsophon P, Roadrangka V, Campbell A (2007) Counting Buttons: demonstrating the Hardy-Weinberg principle. Science in School 6: 30-35.
- Sabeti PC et al. (2006) Positive natural selection in the human lineage. Science312(5780): 1614-20. doi: 10.1126/science.1124309
- Qui sono riassunte in maniera eccellente le varie metodiche utilizzate per studiare la selezione positiva da una prospettiva genomica.
- Sabeti PC et al. (2007) Genome-wide detection and characterization of positive selection in human populations. Nature 449: 913-918. doi: 10.1038/nature06250
- È possibile scaricare l’articolo gratuitamente dal sito di Science in School qui, oppure è necessario registrarsi a Nature al sito: www.nature.com/subscribe
- Questo articolo descrive uno degli approcci di ricerca definita ‘genome-wide’ per individuare la selezione positiva.
- Xue Y et al (2009) Population differentiation as an indicator of recent positive selection in humans: an empirical evaluation. Genetics 183(3): 1065-77. doi:10.1534/genetics.109.107722
- Questo articolo articola una discussione riguardo ad EDAR e altri geni simili. È disponibile gratuitamente su PubMed Central: www.ncbi.nlm.nih.gov/pmc o utilizzando direttamente il link: http://tinyurl.com/26xte2h
Web References
- w1 – Il progetto HapMap è una collaborazione tra scienziati e agenzie di finanziamento del Canada, Cina, Giappone, Nigeria, Regno Unito e Stati Uniti d’America per lo sviluppo di un ente pubblico che potrà aiutare i ricercatori ad individuare geni associati a malattie e importanti nella risposta a terapie farmaceutiche. Vedi il sito: www.hapmap.org
- w2 – Per ulteriori informazioni riguardo il genoma e il progetto Genoma Umano, vedi ‘What is a genome’ (‘che cosa è il genoma’) al sito internet della Libreria Medica Nazionale Statunitense: http://ghr.nlm.nih.gov/handbook/hgp/genome
Review
Nonostante ad oggi si conosca tutta la sequenza del genoma umano, la precisa funzione dei suoi ampi segmenti nonché il come ed il perché le sequenze di DNA siano cambiate nelle popolazioni rimangono argomenti profondamente sconosciti. L’adattamento evolutivo negli uomini è senza dubbio avvenuto ma è molto difficile dimostrarlo. Questo articolo descrive come uno di questi cambiamenti è stato identificato. Esperimenti condotti con topi geneticamente modificati hanno dimostrato la variazione di una singola base di DNA, che porta alla modifica della sequenza aminoacidica della proteina, causa un’alterazione della struttura e della funzione della proteina stessa. Questo in fine risulta in una variazione fenotipica.
Nelle lezioni di scienze, questo articolo può essere utilizzato per approfondire argomenti quali l’utilizzo del codice dei codoni e la loro degenerazione; struttura e funzione proteica e genetica delle popolazioni. Inoltre può essere usato per creare una conoscenza propedeutica utile per affrontare argomenti come le variazioni della popolazione umana o come punto di partenza per approfondire poi le attività dell’Istituto Sanger e il progetto della mappatura del Genoma Umano.
Gli studenti potranno così discutere sul vantaggio evolutivo con riferimenti alla variazione descritta in questo articolo. Questo potrebbe fornire interessanti spunti per una discussione sulla selezione, sulla genetica delle popolazioni e sull’equilibrio Hardy-Weinberg. Per completare questa discussione, c’è un’interessante attività nel numero 6 di Science in School (Pongsophon et al., 2007).
Possibili quesiti di comprensione possono essere:
- Descrivi usando parole tue e dando un esempio, che cosa è uno SNP.
- Spiega il significato di SNPs.
- Quale aminoacido è codificato dalla tripletta di nucleotidi GTT?
- Descrivi quail sono i cambiamenti fenotipici rilevati nei topi geneticamente modificati nella via di segnale di EDAR e suggerisci i modi in cui questi cambiamenti potrebbero essere quantificati.
Shelley Goodman, Regno Unito





