Evolución humana: comprobando las bases moleculares Understand article
Traducido por MariaRosa Quintero Bernabeu. En este segundo artículo, Jarek Bryk describe cómo los científicos investigan a fondo en nuestros genes para probar las bases moleculares de la adaptación evolutiva en humanos.

iStockphoto
El ADN de cada organismo contiene información sobre su historia evolutiva antigua y reciente. Estudiando los patrones y cambios en la secuencia de ADN, comparando secuencias de diferentes individuos o especies- es posible descubrir que les pasó. Podemos saber que genes o fragmentos del genoma es más probable que proporcionaran una ventaja a estos individuos y especies, permitiendo una mejor supervivencia y reproducción (ver el glosario para definiciones de los términos en negrita).
En un artículo anterior (Bryk, 2010), describí algunos ejemplos de estos cambios genéticos beneficiosos para los humanos y otros organismos. Demostrar qué cambios genéticos pueden haber sido beneficiosos es difícil, especialmente en humanos, pero lo que supone un reto todavía mayor es demostrar el mecanismo por el cual estos cambios pudieron llegar a mejorar la supervivencia y la reproducción de los organismos.
En el presente artículo muestro uno de los enfoques que los científicos pueden usar para identificar inicialmente regiones de nuestro genoma que pueden haberayudado a sobrevivir y a reproducirnos, y después probar cómo estas regiones pueden haber supuesto una ventaja para nuestros ancestros.
Una manera de identificar regiones del genoma que pueden ser potencialmente beneficiosas es comparar secuencias de DNA de muchos individuos de diferentes poblaciones. En el caso más simple, si una de estas poblaciones ha estado bajo una presión de selección (por ejemplo alto grado de radiación UV en una región soleada) que no existía en otra población la secuencia de ADN responsable de la correcta adaptación (por ejemplo, color de piel más oscuro) debería ser diferente.
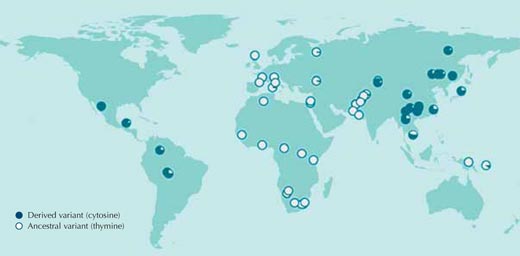
Adaptado con permiso de Macmillan Publishers Ltd: Nature, Sabeti et al. (2007), © 2007
En la gran mayoría de casos, las presiones de selección a las que fueron sometidas las poblaciones en el pasado son desconocidas, así como las secuencias genéticas responsables de estas adaptaciones. Comencemos pues comparando secuencias de ADN entre poblaciones humanas sin hacer suposiciones sobre qué resultados podemos obtener. La figura 1 muestra una comparación de este tipo para un único nucleótido del genoma humano.
Si varios individuos presentan diferentes nucleótidos en una posición concreta de la secuencia de ADN, esta diferencia se llama polimorfismo del nucleótido único (single nucleotide polymorphism SNP, que se pronuncia”‘snip”). Tres millones de estas variantes del genoma humano están catalogadas en la base de datos pública HapMapw1. El SNP que se muestra en la figura 1, rs3827760, se encuentra en dos variantes o alelos. En este punto de la secuencia podemos encontrar dos bases: bien timina (T) bien citosina (C).
Cada círculo en la figura representa una única población, y describe la frecuencia de los dos posibles alelos en dicha población.
El alelo que contiene timina se encuentra presente en todas la muestras africanas y en gran parte se las europeas, pero no en las muestras del este de Asia y americanas, en las que la citosina es más común en esta posición de la secuencia (Sabeti et al., 2007, 2006; Xue et al., 2009).
Si hiciéramos esta comparación con otros tres millones de SNPs que encontramos en el HapMap veríamos que la distribución de las variantes del rs3827760 entre las poblaciones humanas es muy poco usual. Por eso rs3827760 merece una mirada más detallada, a pesar de que la distribución no nos dice nada sobre el potencial beneficio de las variantes (su valor adaptativo) o ni siquiera si son adaptativas. Lo único que sabemos hasta ahora es que, por algún motivo, la timina original que estaba presente en esta posición en las poblaciones humanas ancestrales en África cambió a citosina, y que este cambio se expandió al este de Asia y América. Incluso las estimaciones sobre cuando se produjo este cambio son muy poco precisas: en algún punto entre 1000 y 70000 atrás, todos los individuos del este de Asia presentaban la variante citosina.
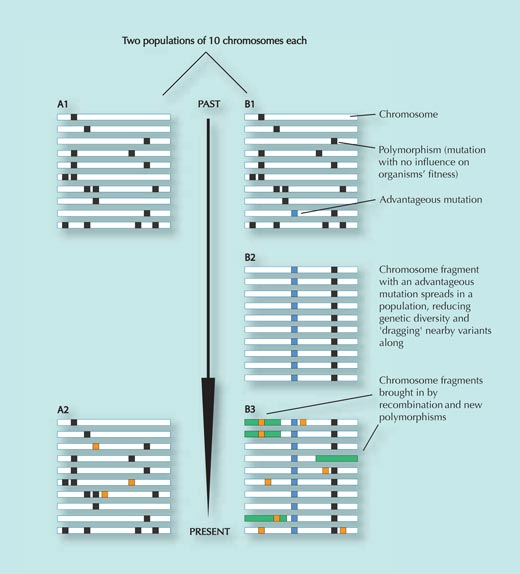
La población A no está sometida a la presión de selección y se mantiene relativamente constante a lo largo del tiempo, además de adquirir algunos cambios genéticos al azar que no afectan a su eficacia biológica inclusiva (los cuadrados naranjas en el panel A2; variantes que reducen la eficacia biológica inclusiva son eliminadas de la población). Comparad los paneles A1 y A2.
Sin embrago, la población B se desplaza hacia un nuevo entorno, donde se enfronta a nuevas presiones selectivas. En este nuevo entorno, un cambio genético particular (el cuadrado azul en el panel B1) proporciona una ventaja a los individuos que lo presentan y se expande rápidamente a toda la población (los individuos que la presentan dejan más descendencia). Las variantes genéticas cercanas al SNP seleccionado se ven arrastradas con él (cuanto más cerca están dos variantes, más pequeña es la posibilidad de que se separen durante la recombinación, cuando partes del ADN son intercambiadas entre los cromosomas paternos y maternos. Ver figura 3).
El resultado de esta rápida expansión de la secuencia de ADN a través de la población es una reducción de la diversidad genética en dicha región. La mayoría de individuos tendrán un SNP ventajoso, así como las variantes genéticas cercanas a él (comparad los paneles B1 y B2). Este proceso se produce rápidamente.
Al cabo de algún tiempo, sin embargo, nuevos cambios genéticos y procesos de recombinación introducen nuevas variantes (rectángulos verdes y cuadrados naranjas en el panel B3). Cuanto más tiempo pasa desde la expansión de la variante seleccionada original, más difícil resulta de detectar, porque el patrón de diversidad reducida (B2) será cubierto en algún momento (B3)
Imagen cortesía de Jarek Bryk
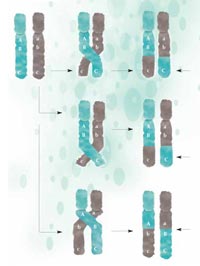
recombinación, partes del
ADN son intercambiadas
entre los cromosomas
maternos (verde) y paternos
(gris) y pasan a la nueva
generación en una nueva
configuración. Cuanto más
cerca se encuentran dos
regiones, menos probable es
que se separen durante la
recombinación. A y B están
más cerca que B y C, por lo
tanto es menos probable que
A y B se separen. Haga clic
sobre la imagen para
ampliarla
Imagen cortesía de Nicola Graf
¿Cómo podemos pues decidir si este patrón apareció debido a una selección positiva (es decir, la citosina confería una ventaja a las poblaciones del este de Asia y de América) o simplemente debido al azar? Para ver si el cambio en el ADN (timina a citosina) fue positivamente seleccionado, hay que echar un vistazo a la secuencia de ADN alrededor del SNP. Si la secuencia de ADN alrededor del rs3827760 fuera similar en todas las poblaciones, no tendríamos ninguna evidencia de que el SNP tuvo un efecto en la eficacia biológica inclusiva de estos organismos. Si, en cambio, una población (por ejemplo, este de Asia) estuvo expuesta a una presión selectiva y el rs3827760 contribuyó al desarrollo de la adaptación a esta presión selectiva, las secuencias de ADN alrededor del SNP serian diferentes entre las diferentes poblaciones. Para entender por qué esto es así, veamos la figura 2.
Cuando se comparan las secuencias de ADN alrededor de rs3827760, se hace evidente que la diversidad alrededor de las variantes de citosina en las poblaciones del este de Asia es mucho más baja que la diversidad alrededor de las variantes de tiamina en poblaciones de África y Europa (las poblaciones de América no fueron estudiadas). Estos resultados sugieren que la selección positiva fue responsable de la expansión de la variante de citosina en las poblaciones del este de Asia. Pero ¿fue realmente seleccionado, este SNP? ¿Tiene esto algún efecto?
No todos los cambios en las secuencias de ADN tienen un efecto en las secuencias de proteínas: la mayoría de los SNPs catalogados en la base de datos HapMap se encuentran o bien en partes del genoma no codificantes (es decir, entre genes) o son sinónimos, es decir, están localizados en regiones codificantes del genoma pero no producen cambios en la secuencia de la proteína codificada (see Figure 4).
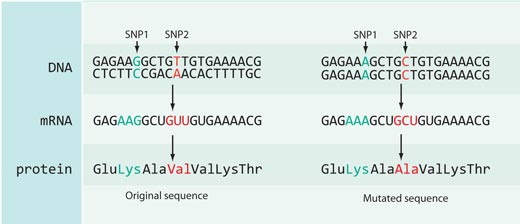
Imagen cortesía de Jarek Bryk
En el caso de rs3827760 estamos de suerte, porque está localizado en la parte codificante del gen, hacia el final de un gen llamado EDAR, el cual está involucrado en el desarrollo de los folículos capilares, las glándula sudoríparas y los dientes. Además, el cambio de timina a citosina en la secuencia de ADN resulta en un cambio en la secuencia de la proteína: los africanos y los europeo (que tienen la variante de SNP con timina) tienen el aminoácido valina en la posición 370 de la proteína, mientras que los asiáticos del este y los americanos (con el nucleótido citosina) presentan el aminoácido alanina. Esta parte de la proteina está relacionada con interacciones con otras proteínas, y se sabe que mutaciones en esta zona están relacionadas con displasias ectodérmicas (desarrollo anormal de los dientes, el pelo y las glándulas sudoríparas) en humanos y en ratas (ver figura 5). Este hecho sugiere que el cambio en el aminoácido en la posición 370 puede no tan sólo cambiar la secuencia de la proteína sino cómo esta se comporta, afectando a las características físicas del propio organismo.
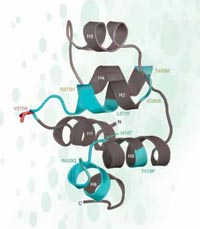
hipotética de una parte de la
proteína EDAR. Las
mutaciones marcadas en
verde causan displasia
ectodérmica en humanos. El
SNP putativo seleccionado se
presenta en rojo
Adaptado con permiso de
Macmillan Publishers Ltd:
Nature, Sabeti et al. (2007), ©
2007
Para comprobar si el cambio en la secuencia de la proteína afecta realmente a su función, debemos fijarnos en experimentos en la ruta bioquímica en la que la proteína EDAR participa, una serie de reacciones que están involucradas en el desarrollo de folículos capilares, glándulas sudoríparas y dientes. Cuando estas reacciones se realizaron en el laboratorio, se encontró que la variante que contenía alanina (que se encuentra en la poblaciones de América y del este de Asia, y que está codificada por citosina en la variante del SNP) volvía la ruta bioquímica más activa que la variante que contenía valina (presente en las poblaciones africana y europea, y codificada por una timina en la variante SNP). Esto está relacionado con las comparaciones entre las estructuras del pelo, que mostraron que las personas que presentan la variante alanina tienen un pelo más grueso que las personas que presentan la variante valina. Para obtener una demostración más directa, se modificaron genéticamente ratones para aumentar la actividad de la ruta de EDAR. Estos ratones presentaban un pelo visiblemente más grueso, así cómo glándulas salivares más grandes, que los ratones con una actividad EDAR normal (Chunyan et al., 2008; Chang et al., 2009).
Todos estos resultados sugieren que las dos variantes del SNP (que contienen bien timina bien citosina) pueden afectar tanto a la estructura como a la función de la proteína EDAR, y pueden llevar a diferencias físicas en humanos: diferencias en el grosor del cabello y, potencialmente, en la talla de las glándulas salivales.
Las diferencias en las secuencias de ADN que podemos observar hoy en día son archivos históricos de experimentos naturales, y sólo podemos especular sobre cuales de las presiones selectivas a las que las poblaciones asiáticas y americanas fueron sometidas fueron responsables de la dispersión del alelo citosina. Pero la combinación de estudios genéticos, experimentos de laboratorio y modelos animales hace posible examinar las diferentes hipótesis sobre el papel funcional de las diferencias genéticas entre poblaciones o especies. Utilizando estas aproximaciones podemos descubrir las bases moleculares de adaptaciones pasadas en nuestros ancestros y en otros organismos, destacando cómo nos adaptamos constantemente a los cambios del entorno.
Glosario
Valor adaptativo: un rasgo tiene valor adaptativo si permite al individuo sobrevivir y reproducirse en un entorno determinado mejor que otros individuos que no presentan este rasgo. De manera más formal, un rasgo se considera adaptativo si aumenta la eficacia biológica inclusiva.
Eficacia biológica inclusiva: este es un término difícil de definir que proviene de la biología evolutiva y la genética de poblaciones. Describe la descendencia promedio en una generación que está asociada con un genotipo comparada con la de otro genotipo en la población. Por lo tanto los genotipos que producen más descendencia tienen una eficacia biológica adaptativa más otra.
Genoma: cantidad total de ADN de una célula. Normalmente en eucariotas se considera ADN nuclear, por oposición a ADN mitocondrial o plásmido. Para más información, ver ‘What is a genome’ («¿Qué es un genoma?») en la en la página web de la Biblioteca Nacional de Medicina de los Estados Unidos (US National Library of Medicine website)w2.
Selección positiva: la selección natural es uno de los mecanismos de la evolución, que describe las diferencias de supervivencia y reproducción de los individuos en un entorno determinado. La selección natural se considera positiva cuando favorece determinados rasgos que ayudan al individuo que los presenta a sobrevivir y reproducirse mejor que otros individuos.
Presión de selección: una característica del entorno (por ejemplo temperatura, presencia de parásitos, predadores o agresión por parte de otros miembros de la especie) que impone diferencias en la supervivencia y reproducción de los individuos.
SNP: polimorfismo de un único nucleótido, o letra en la secuencia de ADN que varía entre individuos. Se pronuncia snip.
References
- Bryk J (2010) Selección natural a nivel molecular. Science in School 14: 58-62.
- Chang SH et al. (2009) Enhanced EDAR signalling has pleiotropic effects on craniofacial and cutaneous glands (El aumento de la actividad en la ruta de EDAR tiene efectos pleiotrópicos en las glándulas craneofaciales y cutáneas). PLoS ONE4(10): e7591. doi: 10.1371/journal.pone.0007591
- Esta artículo describe el fenotipo de varias glándulas de ratones con aumento de actividad en la ruta de EDAR, y especula sobre que rasgos pueden haber sido seleccionados positivamente en la historia humana. El artículo es de libre acceso en la página web de la revista: www.plosone.org
- Chunyan M et al. (2008) Enhanced ectodysplasin-A receptor (EDAR) signaling alters multiple fiber characteristics to produce the East Asian hair form (El aumento en la señalización del receptor de ectodysplasin-A (EDAR) altera múltiples características de fibras para producir el tipo de pelo del este de Asia).Human Mutation 29(12): 1405-1411. doi: 10.1002/humu.20795
- Este artículo explica de manera detallada experimentos in vitro de EDAR y ratones transgénicos, con fotografías e imágenes muy interesantes.
- Pongsophon P, Roadrangka V, Campbell A (2007) Counting Buttons: demonstrating the Hardy-Weinberg principle (Contando botones: demostrando el principio de Hardy-Weinberg). Science in School 6: 30-35.
- Sabeti PC et al. (2006) Positive natural selection in the human lineage (Selección positiva natural en el linaje humano). Science 312(5780): 1614-20. doi:10.1126/science.1124309
- Este artículo presenta un resumen de varios métodos usados para investigar la selección positiva desde una perspectiva genómica.
- Sabeti PC et al. (2007) Genome-wide detection and characterization of positive selection in human populations (Detección y caracterización a nivel genómico de la selección positive en poblaciones humanas). Nature 449: 913-918. doi:10.1038/nature06250
- El artículo puede ser descargado gratuitamente desde Science in School aquí, o bien suscribiéndose a Nature hoy: www.nature.com/subscribe
- Este artículo describe una de las aproximaciones a las investigaciones de selecciones positivas a nivel genómico.
- Xue Y et al (2009) Population differentiation as an indicator of recent positive selection in humans: an empirical evaluation (Diferenciación de poblaciones como indicador de una selección positiva en humanos reciente: una evaluación empírica). Genetics 183(3): 1065-77. doi: 10.1534/genetics.109.107722
- Este artículo contiene una discusión sobre EDAR y otros genes similares. Se encuentra disponible gratuitamente a través de PubMed Central:www.ncbi.nlm.nih.gov/pmc o usando directamente el enlace:http://tinyurl.com/26xte2h
Web References
- w1 – El proyecto HapMap es una asociación de científicos y agencias de financiación de Canadá, China, Japón, Nigeria, el Reino Unido y EUA que tiene como objetivo desarrollar un recurso público que pueda ayudar a los investigadores a encontrar genes asociados con enfermedades humanas y su respuesta a medicamentos. Consultad: www.hapmap.org
- w2 – Para obtener más información sobre genomas y el proyecto Human Genome, consultad “¿Qué es un genoma?” en la página web de la Biblioteca Nacional de Medicina de Estados Unidos (US National Library of Medicine):http://ghr.nlm.nih.gov/handbook/hgp/genome
Review
A pesar de nuestro conocimiento de la secuencia del genoma humano, todavía se desconocen la función exacta de algunos de los segmentos que contiene y cómo y porqué las secuencias de ADN son diferentes entre poblaciones. Obviamente se ha producido adaptación evolutiva en humanos pero es muy difícil de demostrar. Este artículo describe cómo se pueden identificar este tipo de cambios. Experimentos con ratones genéticamente modificados han demostrado cómo el cambio en una sola base del ADN que cambia la secuencia de aminoácidos de la proteína puede llevar a una alteración de la función y la estructura de dicha proteína. Este cambio puede conllevar variaciones fenotípicas.
En las clases de ciencias, este artículo puede ser utilizado para ilustrar los temas de uso y degeneración de codones y función y estructura de proteínas. Puede también utilizarse como lectura complementaria sobre variaciones entre poblaciones humanad o como punto de partida para una investigación sobre el Instituto Sanger y el Human Genome Mapping Project.
Los alumnos pueden discutir sobre ventajas evolutivas, haciendo referencia en particular a la variación descrita en este artículo. Esto podría llevar a una discusión sobre selección, genética de poblaciones y equilibrio de Hardy-Weinberg. Para completar esta discusión existe una excelente actividad en el número 6 de Science in School (Pongsophon et al., 2007).
Algunas preguntas de comprensión pueden ser:
- En tus propias palabras define que es un SNP. Da un ejemplo diferente del del texto.
- Explica la importancia de los SNP.
- ¿Para qué aminoácido codifica el triplete GTT?
- Describe los cambios que se encontraron en los ratones genéticamente modificados en la ruta de EDAR. Sugiere diferentes maneras para cuantificar los cambios observados.
Shelley Goodman, Reino Unido





