De evolutie van de mens: de moleculaire basis testen Understand article
Vertaald door Carolien de Kovel. In het tweede van twee artikelen beschrijft Jarek Bryk hoe wetenschappers diep in onze genen graven om de moleculaire basis te testen van evolutionaire aanpassing in de mens.

/iStockphoto
Het DNA van elk organisme bevat informatie over zijn recente en vroege evolutionaire geschiedenis. Door de patronen en veranderingen in de DNA-volgorde te bestuderen – de sequenties tussen verschillende individuen of tussen verschillende soorten te vergelijken – kunnen we ontdekken wat er met ze gebeurd is. We kunnen uitvinden welke genen or welke fragmenten van het genoom waarschijnlijk een voordeel hebben geboden aan de individuen en aan de soorten die ze hadden, ofwel bijdroegen aan een betere overleving en reproductie (zie de woordenlijst voor de vetgedrukte termen).
In een eerder artikel (Bryk, 2010), heb ik een aantal voorbeelden beschreven van zulke gunstige genetische veranderingen in mensen en andere organismen. Het is moeilijk om aan te tonen welke genetische veranderingen mogelijk gunstig zijn geweest, vooral bij mensen, maar een nog grotere uitdaging is om aan te tonen welk mechanisme bij deze veranderingen de overleving en de voortplanting van het organisme heeft verbeterd.
In dit artikel presenteer ik een van de aanpakken die wetenschappers kunnen gebruiken om eerst regio’s in ons genoom te identificeren die mogelijk hebben bijgedragen aan overleving en voortplanting, en om dan te testen hoe deze regio’s onze voorouders tot voordeel zijn geweest.
Een van de manieren om mogelijk gunstige regio’s in ons genoom te identificeren is om eenvoudigweg DNA-sequenties van veel individuen uit verschillende populaties te vergelijken. In een heel simpel scenario, als één van deze populaties onder selectiedruk heeft gestaan (bijvoorbeeld hoge UV-straling in een zonnig gebied) die niet aanwezig was in de andere populaties, dan zou de DNA-sequentie die verantwoordelijk is voor de aanpassing (bijvoorbeeld, donkerder huid) anders moeten zijn.
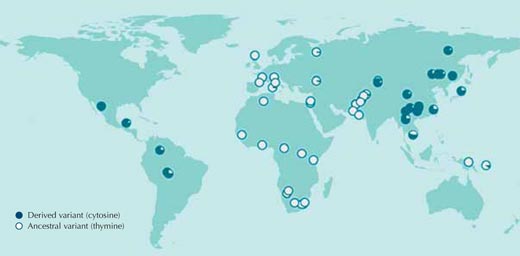
Aangepast met toestemming van Macmillan Publishers Ltd Sabeti et al. (2007), © 2007
In de overgrote meerderheid van de gevallen, echter, weten we niet aan wat voor selectiedrukken de populaties onderhevig zijn geweest in het verleden, of wat voor genetische sequenties verantwoordelijk zijn voor de aanpassingen. Laten we daarom beginnen DNA-sequenties tussen menselijke populaties te vergelijken zonder aannames over wat we zouden kunnen vinden. Figuur 1 laat zo’n vergelijking zien, voor een enkele nucleotide in het menselijk genoom.
Als individuen verschillende nucleotiden (DNA-letters) hebben op een bepaalde positie in de DNA-sequentie, dan noemen we dit een single nucleotide polymorphism (SNP, uitgesproken als ‘snip’); drie miljoen van zulke varianten in het humane genoom staan opgetekend in de publiek beschikbare HapMap databankw1. The SNP in Figuur 1, rs3827760, wordt aangetroffen met twee varianten of allelen: op dat punt in de sequentie wordt zowel een thymine (T) aangetroffen als een cytosine (C).
Elke rondje in de figuur stelt een aparte populatie voor en laat de frequentie in die populatie zien van de twee verschillende allelen.
Het thymine-allel komt voor in alle Afrikaanse en de meeste Europese monsters, maar is vrijwel geheel afwezig in Oost-Azië en in de Nieuwe Wereld, waar cytosine de meest voorkomende variant op die positie is (Sabeti et al., 2007, 2006; Xue et al., 2009).
Als we dezelfde vergelijking zouden uitvoeren voor al de drie miljoen SNPs uit de HapMap, dan zouden we zien dat de verdeling van de rs3827760-varianten over de menselijke populaties erg ongebruikelijk is. Daarom verdient rs3827760 beslist nader bekeken te worden, ook al vertelt de verdeling ons niets over het mogelijk gunstige effect van de varianten (hun aanpassingswaarde), of zelfs maar of ze überhaupt van invloed zijn op aanpassing. Alles wat we op dit moment weten is dat om de een of andere reden het thymine dat oorspronkelijk aanwezig was in de voorouderlijke populaties in Afrika veranderd is in een cytosine en dat deze verandering zich heeft uitgebreid onder Oost-Aziaten en oorspronkelijke bewoners van het Amerikaanse continent. Zelfs de schatting van wanneer dit gebeurde is niet erg precies: ergens tussen 1,000 en 70,000 jaar geleden hadden alle inwoners van Oost-Azië de cytosine-variant.
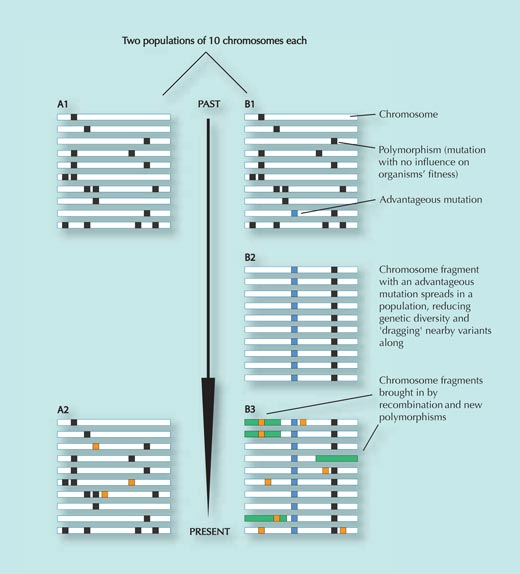
Populatie A ondergaat geen positieve selectie en blijft vrijwel onveranderd over de tijd, afgezien van het verkrijgen van enkele toevallige genetische veranderingen die geen effect op fitness hebben (oranje blokjes in deel A2; varianten die een negatief effect hebben op fitness worden uit de populatie verwijderd) – vergelijk delen A1 en A2.
Populatie B verhuist naar een nieuwe omgeving, waar ze nieuwe selectiedrukken ervaart. In dat nieuwe milieu verleent een bepaalde genetische verandering (blauwe blokje in deel B1) een voordeel aan de individuen die de variatie hebben en de variatie verspreidt zich snel in de populatie (individuen met de variatie krijgen meer overlevende nakomelingen). De genetische variaties dicht bij het geselecteerde polymorfisme worden meegesleept (hoe dichter bij elkaar de varianten zijn, hoe kleiner de kans dat ze door recombinatie worden gescheiden als delen van paternale en maternale chromosomen worden uitgewisseld in de meiose – zie Figuur 3).
Het resultaat van deze snelle verspreiding van een DNA-sequentie in een populatie is een reductie van de genetische diversiteit in die regio; de meeste individuen zullen de gunstige SNP dragen, evenals de genetisch varianten die er dicht bij liggen (vergelijk delen B1 en b2). Dit proces verloopt snel.
Na enige tijd, echter, introduceren nieuwe genetische veranderingen en overkruisingen nieuwe varianten (groene rechthoekjes en oranje vierkantjes in deel B3). Hoe langer het geleden is dat de oorspronkelijke gunstige variant zich verspreid heeft, hoe moeilijker dat nog te detecteren is, omdat het patroon van gereduceerde diversiteit (B2) uiteindelijk gemaskeerd raakt (B3)
Figuur met dank aan Jarek Bryk
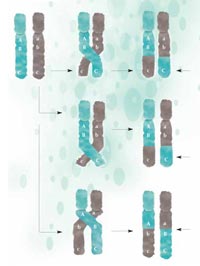
worden delen van het DNA
uitgewisseld tussen het
maternale (groene) en
paternale (grijze)
chromosoom en doorgegeven
aan de volgende generatie in
een nieuwe samenstelling.
Hoe dichter bij elkaar twee
regio’s zijn op het
chromosoom, hoe minder
waarschijnlijk het is dat ze
bij recombinatie gescheiden
worden: A en B zijn dichter
bij elkaar dan B en C en dus
is het minder waarschijnlijk
dat ze gescheiden raken. Klik
op de afbeelding om te
vergroten.
Figuur met dank aan Nicola Graf
Hoe kunnen we dan uitmaken of dit patroon het gevolg is van positieve selectie (cytosine gaf een voordeel in Oost-Azië en de Nieuwe Wereld) of gewoon een toevalstreffer is? Om te zien of de DNA-verandering (van thymine naar cytosine) onder positieve selectie stond, kijken we naar de DNA-sequentie rondom de SNP. Als de DNA-sequentie rondom rs3827760 vergelijkbaar is in alle populaties, dan zouden we geen bewijs hebben dat de SNP een effect had op de fitness. van het organisme. Als echter één populatie (Oost-Azië bijvoorbeeld) onder selectie stond en rs3827760 droeg bij aan de ontwikkeling van een aanpassing aan deze selectiedruk, dan zouden de DNA-sequenties rondom de SNP verschillen. Om te begrijpen waarom dat zo is, zie Figuur 2.
Als de DNA-sequenties rond rs3827760 vergeleken worden, wordt het duidelijk dat de diversiteit rond de cytosine-varianten in de Oost-Aziatische populaties inderdaad veel lager is dan de diversiteit rond de thymine-varianten die gevonden worden in de Afrikaanse en Europese populaties (Amerika werd niet getest). Dit suggereert dat positieve selectie verantwoordelijk was voor het verspreiden van de cytosine-variant in de Oost-Aziatische populaties. Maar was het echt deze SNP waarop geselecteerd werd – doet die SNP wel iets?
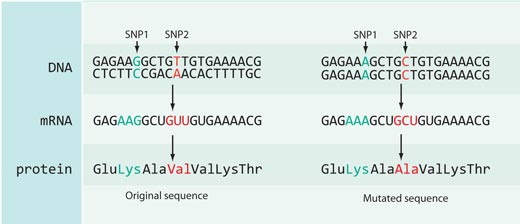
Niet alle veranderingen in de DNA-sequentie hebben een effect op de eiwit-sequenties: de meeste SNPs die in de HapMap-databank staan zijn gelegen in de niet-coderende delen van het genoom (bijv. tussen genen in) of ze zijn synoniem – dat betekent, ze zitten in het coderende gedeelte van het genoom, maar veranderen niet de eiwit-sequentie (zie figuur 4).
Figuur met dank aan Jarek Bryk
In het geval van rs3827760 hebben we geluk, omdat die zich bevindt in het coderende gedeelte van een gen – te weten achter in een gen dat EDAR heet en dat betrokken is bij de ontwikkeling van haarfollikels, zweetklieren en tanden. Bovendien verandert de thymine-cytosine DNA-sequentie-verandering de eiwitsequentie: Afrikanen en Europeanen (die de SNP-variant met thymine hebben) hebben het aminozuur valine op positie 370 van het eiwit, terwijl de Oost-Aziaten en de Amerikaanse indianen (met de cytosine-variant) het aminozuur alanine hebben. Dit gedeelte van het eiwit is betrokken bij interacties met andere eiwitten en mutaties (veranderingen) hier, zo is bekend, zorgen voor ectodermale dysplasieën – abnormale ontwikkeling van tanden, haar en zweetklieren – in mens en in muis (zie Figuur 5). Dit suggereert sterk dat een aminozuurverandering op positie 370 niet alleen de sequentie van het eiwit verandert, maar ook kan veranderen hoe het eiwit zich gedraagt, en daardoor de fysieke kenmerken van het organisme zelf kan veranderen.
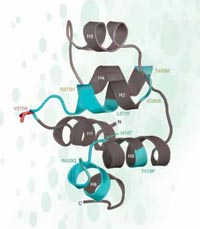
structuur van een deel van
het EDAR-eiwit. Mutaties in
groen veroorzaken
ectodermale dysplasie in
mensen. De verondersteld
geselecteerde SNP is in rood
aangegeven
Aangepast met toestemming
van Macmillan Publishers Ltd:
Nature, Sabeti et al. (2007), ©
2007
Om te zien of de verandering in de eiwitsequentie een effect heeft op de functie, nemen we onze toevlucht tot experimenten met de biochemische route waar het EDAR-eiwit een rol in speelt: een serie van reacties die betrokken zijn bij de ontwikkeling van haarfollikels, zweetklieren en tanden. Toen deze reacties in het laboratorium werden uitgevoerd, werd gevonden dat de alanine-variant van het eiwit (gevonden in Oost-Aziaten en Amerikaanse indianen, gecodeerd door de cytosine-SNP-variant) de reactieketen actiever maakte dan de valine-variant (gevonden in Afrikanen en Europeanen, gecodeerd door de thymine-SNP-variant). Dit past goed bij vergelijkingen van de haarstructuur die laten zien dat mensen met de alanine-variant dikker haar hebben dan de mensen met de alanine-variant. Voor een directer bewijs werden muizen genetisch gemodificeerd om de activiteit van de EDAR-reactieketen te verhogen. De muizen hadden een zichtbaar dichtere vacht met dikker haar, evenals grotere speekselklieren dan muizen met normale EDAR-activiteit (Chunyan et al., 2008; Chang et al., 2009).
Alles bij elkaar suggereren deze bevindingen dat de twee SNP-varianten (thymine of cytosine) zowel de structuur als de functie van het EDAR-eiwit veranderen en kunnen leiden tot fysieke verschillen in mensen: verschillen in haardikte en, mogelijk, de grootte van de speekselklieren.
De verschillen in de DNA-sequenties die we nu zien zijn historische archieven van natuurlijke experimenten, en we kunnen alleen maar speculeren over de selectiedrukken waar de Aziatische en Amerikaanse bevolking aan hebben blootgestaan, die de verspreiding van de cytosine-variant bevorderd hebben. Maar de combinatie van genomisch onderzoek, laboratoriumexperimenten en diermodellen maken het mogelijk om hypothesen te testen over de functionele rollen die genetische verschillen tussen populaties of soorten spelen. Met behulp van deze aanpakken kunnen we de moleculaire basis ontrafelen van aanpassingen uit het verleden in onze voorouders en andere organismen, waarbij duidelijk wordt hoe we ons aanpassen aan een steeds veranderende omgeving.
Woordenlijst
Aanpassingswaarde: een kenmerk heeft een aanpassingswaarde als het het individu in staat stelt om beter te overleven en te reproduceren in een bepaald milieu dan individuen die dit kenmerk niet bezitten. Formeler, een kenmerk wordt als adaptief beschouwd als het fitness verhoogt.
Fitness: een moeilijk te definiëren formele term uit de evolutionaire biologie en populatiegenetica; het beschrijft het gemiddelde aantal nakomelingen over één generatie dat is geassocieerd met het ene genotype in een populatie vergeleken met een ander genotype. Het genotype dat meer nakomelingen produceert heeft een hogere fitness.
Genoom: het totale DNA van een organisme. Meestal bedoeld in eukaryoten als het totale nucleaire DNA, in tegenstelling tot alle DNA inclusief dat in mitochondriën of plastiden. Voor meer informatie, zie ‘What is a genome’ op de website van de US National Library of Medicinew2.
Positieve selectie: natuurlijke selectie is een van de mechanismen van evolutie; het beschrijft de verschillende overleving en voortplanting van individuen in een bepaal milieu. Natuurlijke selectie wordt ‘positief’ genoemd als het bepaalde kenmerken bevordert, kenmerken die individuen helpen om beter te overleven en voort te planten dan andere.
Selectiedruk: een eigenschap van het milieu (bijvoorbeeld temperatuur; aanwezigheid van parasieten; predatie of agressie van de eigen soort) die zorgt voor verschillende overleving en reproductie van individuen.
SNP: een ‘single nucleotide polymorphism’ (enkele nucleotide polymorfisme), of een enkele letter in de DNA-sequentie die verschilt tussen individuen. Uitgesproken als ‘snip’.
References
- Bryk J (2010) Natural selection at the molecular level. Science in School 14: 58-62.
- Chang SH et al. (2009) Enhanced EDAR signalling has pleiotropic effects on craniofacial and cutaneous glands. PLoS ONE 4(10): e7591. doi:10.1371/journal.pone.0007591
- Dit artikel beschrijft het fenotype van verschillende klieren in muizen met verhoogd EDAR-signaal, en speculeert welke kenmerken onder positieve selectie kunnen hebben gestaan in de geschiedenis van de mens. Het artikel kan gratis worden opgehaald van de website: www.plosone.org
- Chunyan M et al. (2008) Enhanced ectodysplasin-A receptor (EDAR) signaling alters multiple fiber characteristics to produce the East Asian hair form. Human Mutation 29(12): 1405-1411. doi: 10.1002/humu.20795
- Het artikel beschrijft in detail in vitro-studies van EDAR en de transgene muizen, met hele mooie foto’s en plaatjes.
- Pongsophon P, Roadrangka V, Campbell A (2007) Counting Buttons: demonstrating the Hardy-Weinberg principle. Science in School 6: 30-35.
- Sabeti PC et al. (2006) Positive natural selection in the human lineage. Science312(5780): 1614-20. doi: 10.1126/science.1124309
- Dit is een uitmuntend overzicht van verschillende methoden die worden gebruikt om positieve selectie te onderzoeken vanuit een genomisch perspectief.
- Sabeti PC et al. (2007) Genome-wide detection and characterization of positive selection in human populations. Nature 449: 913-918. doi: 10.1038/nature06250
- Download het artikel hier gratis, of abonneert u zich op Nature via: www.nature.com/subscribe
- Dit artikel beschrijft een van de manieren om genoomwijd te zoeken naar signalen van positieve selectie.
- Xue Y et al (2009) Population differentiation as an indicator of recent positive selection in humans: an empirical evaluation. Genetics 183(3): 1065-77. doi:10.1534/genetics.109.107722
- Dit artikel bevat een discussie over EDAR en vergelijkbare genen. Het kan gratis worden opgehaald via Pubmed Central: www.ncbi.nlm.nih.gov/pmcof via de directe link: http://tinyurl.com/26xte2h
Web References
- w1 –Het HapMap-project is een partnerschap van wetenschappers en fondsen uit Canada, China, Japan, Nigeria, Groot-Brittannië en de VS om een algemene hulpbron te ontwikkelen die onderzoekers kan helpen genen te vinden die geassocieerd zijn met humane ziekten en met de reactie op medicijnen. Zie:www.hapmap.org
- w2 – Voor meer informatie over genomen en het Humane GenoomProject, zie ‘What is a genome’ op de website van de US National Library of Medicine:http://ghr.nlm.nih.gov/handbook/hgp/genome
Review
Ondanks al onze kennis van de sequentie van het humane genoom, weten we nog weinig over de precieze functie van grote segmenten ervan en over hoe en waarom DNA-sequenties veranderd zijn in populaties. Evolutionaire aanpassing in mensen heeft zondermeer plaatsgevonden, maar het is erg moeilijk aan te tonen. Dit artikel beschrijft hoe één zo’n verandering geïdentificeerd is. Experimenten met genetisch gemodificeerde muizen hebben laten zien hoe een verandering van een enkele base in het DNA, die op zijn beurt de aminozuursequentie van het eiwit verandert, leidt tot een verandering in de structuur of de functie van het eiwit. Dit kan resulteren in fenotypische variatie.
In wetenschapslessen kan dit artikel worden gebruikt als onderwerpen aan de orde zijn zoals codongebruik en degeneratie, eiwitstructuur en –functie, en populatiegenetica. Het kan ook worden gebruikt als achtergrondmateriaal over variatie in de menselijke populatie of als beginpunt voor een studie van het Sangerinstituut en het Human Genome Mapping Project.
De studenten kunnen evolutionair voordeel bespreken, met betrekking tot de specifieke variatie die in het artikel beschreven wordt. Dit kan leiden tot een discussie over selectie, populatiegenetica en Hardy-Weinberg-evenwicht. Om de discussie volledig te maken is er een uitmuntende activiteit in uitgave 6 van Science in School (Pongsophon et al., 2007).
Geschikte vragen naar begrip zouden zijn:
- Beschrijf in je eigen woorden wat een SNP is en geef een voorbeeld dat niet in de tekst wordt besproken.
- Leg het belang van SNPs uit.
- Voor welk aminozuur codeert het triplet GTT?
- Beschrijf de veranderingen die gevonden werden in de muizen met gemodificeerde EDAR-route, een bedenk manieren waarop de geobserveerde veranderingen gekwantificeerd zouden kunnen worden.
Shelley Goodman, GB





