Evolució humana: comprovant les bases moleculars Understand article
Traduït per MariaRosa Quintero Bernabeu. En aquest segon article, Jarek Bryk descriu com els científics investiguen a fons en els nostres gens per provar les bases moleculars de l’adaptació evolutiva en humans.

iStockphoto
L’ADN de cada organisme conté informació sobre la seva història evolutiva antiga i recent. Estudiant els patrons i canvis en la seqüència d’ADN, comparant seqüències de diferents individus o espècies, és possible descobrir que els va passar. Podem saber quins gens o fragments del genoma és més probable que proporcionessin un avantatge a aquests individus i espècies, permetent una millor supervivència i reproducció (vegeu el glossari per definicions dels termes en negreta).
En un article anterior (Bryk, 2010), vaig descriure alguns exemples d’aquests canvis genètics beneficiosos per als humans i altres organismes. Demostrar quins canvis genètics poden haver estat beneficiosos és difícil, especialment en humans, però el que suposa un repte encara més gran és demostrar el mecanisme pel qual aquests canvis van poder arribar a millorar la supervivència i la reproducció dels organismes.
En aquest article mostro un dels enfocaments que els científics poden utilitzar per identificar inicialment regions del nostre genoma que poden haver-nos ajudat a sobreviure i a reproduir-nos, i després provar com aquestes regions poden haver suposat un avantatge per als nostres avantpassats.
Una manera d’identificar regions del genoma que poden ser potencialment beneficioses és comparar seqüències de DNA de molts individus de diferents poblacions. En el cas més simple, si una d’aquestes poblacions ha estat sota una pressió de selecció (per exemple alt grau de radiació UV en una regió assolellada) que no existia en una altra població, la seqüència d’ADN responsable de la correcta adaptacó (per exemple, color de pell més fosc) hauria de ser diferent.
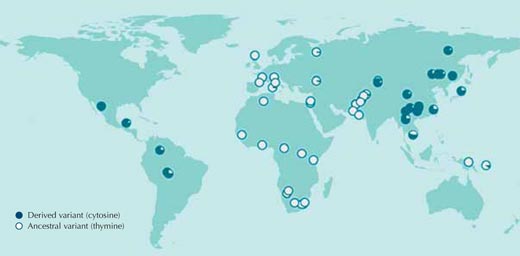
Adaptat amb permís de Macmillan Publishers Ltd: Nature, Sabeti et al. (2007), © 2007
En la gran majoria de casos, les pressions de selecció a què van ser sotmeses les poblacions en el passat són desconegudes, així com les seqüències genètiques responsables d’aquestes adaptacions. Comencem doncs comparant seqüències d’ADN entre poblacions humanes sense fer suposicions sobre quins resultats podem obtenir. La figura 1 mostra una comparació d’aquest tipus per un únic nucleòtid del genoma humà.
Si diversos individus presenten diferents nucleòtids en una posició concreta de la seqüència d’ADN, aquesta diferència es diu polimorfisme del nucleòtid únic (single nucleotide Polymorphism SNP, que es pronuncia “‘snip”). Hi ha tres milions d’aquestes variants del genoma humà catalogades a la base de dades pública HapMapw1. L’SNP que es mostra a la figura 1, rs3827760, es troba en dues variants o al·lels. En aquest punt de la seqüència podem trobar dues bases: bé timina (T) bé citosina (C).
Cada cercle a la figura representa una única població, i descriu la freqüència dels dos possibles al·lels en aquesta població.
L’al·lel que conté timina es troba present en totes les mostres africanes i en gran part de les europees, però no en les mostres de l’est d’Àsia i americanes, en les quals la citosina és més comú en aquesta posició de la seqüència (Sabeti et al., 2007, 2006; Xue et al., 2009).
Si féssim aquesta comparació amb els altres tres milions d’SNPs que trobem al HapMap veuríem que la distribució de les variants del rs3827760 entre les poblacions humanes és molt poc usual. Per això rs3827760 mereix un cop d’ull més de prop, tot i que la distribució no ens diu res sobre el potencial benefici de les variants (el seu valor adaptatiu), o ni tan sols si són adaptatives. L’únic que sabem fins ara és que, per algun motiu, la timina original que estava present en aquesta posició en les poblacions humanes ancestrals a Àfrica va canviar a citosina, i que aquest canvi es va expandir a l’est d’Àsia i Amèrica. Fins i tot les estimacions sobre quan es va produir aquest canvi són molt poc exactes: en algun punt entre 1000 i 70000 enrere, tots els individus de l’est d’Àsia presentaven la variant citosina.
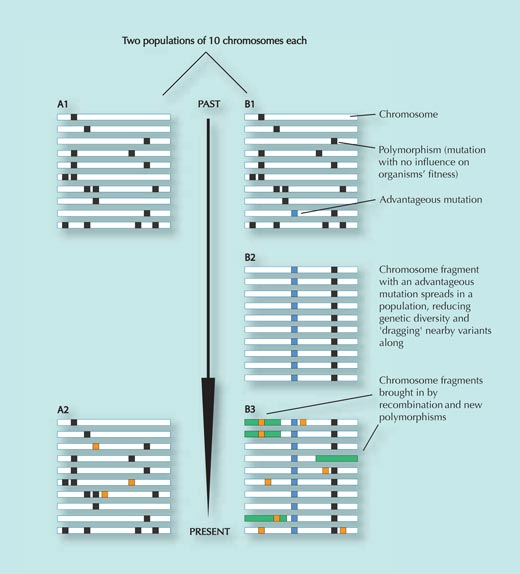
La població A no està sotmesa a la pressió de selecció i es manté relativament constant al llarg del temps, a més d’adquirir alguns canvis genètics a l’atzar que no afecten la seva eficàcia biològica inclusiva (els quadrats taronges al panell A2; variants que redueixen l’eficàcia biològica inclusiva són eliminades de la població). Compareu els panells A1 i A2.
Malgrat tot, la població B es desplaça cap a un nou entorn, on s’enfronta a noves pressions selectives. En aquest nou entorn, un canvi genètic particular (el quadrat blau en el panell B1) proporciona un avantatge als individus que el presenten i s’expandeix ràpidament a tota la població (els individus que la presenten deixen més descendència). Les variants genètiques properes al SNP seleccionat es veuen arrossegades amb ell (com més a prop estan dues variants, més petita és la possibilitat que es separin durant la recombinació, quan parts de l’ADN són intercanviades entre els cromosomes paterns i materns – Veure figura 3).
El resultat d’aquesta ràpida expansió de la seqüència d’ADN a través de la població és una reducció de la diversitat genètica en aquesta regió. La majoria d’individus tindran un SNP avantatjós, així com les variants genètiques properes a ell (compareu els panells B1 i B2). Aquest procés es produeix ràpidament.
Al cap d’un temps, però, els canvis genètics i processos de recombinació introdueixen noves variants (rectangles verds i quadrats taronges en el panell B3). Com més temps passa des de l’expansió de la variant seleccionada original, més difícil resulta de detectar, perquè el patró de diversitat reduïda (B2) serà cobert en algun moment (B3)
Imatge cortesia de Jarek Bryk
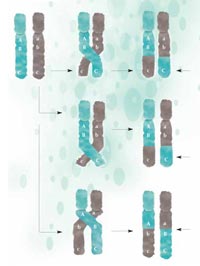
recombinació, parts de l’ADN
són intercanviades entre els
cromosomes materns (verd) i
paterns (gris) i passen a la
nova generació en una nova
configuració. Com més a prop
es troben dues regions,
menys probable és que es
separin durant la
recombinació. A i B estan
més a prop que B i C, per tant
és menys probable que A i B
es separin. Fer clic sobre la
imatge per ampliar-la
Imatge cortesia de Nicola Graf
Com podem doncs decidir si aquest patró va aparèixer a causa ’d’una selecció positiva (és a dir, la citosina conferia un avantatge a les poblacions de l’est d’Àsia i d’Amèrica) o simplement causa de l’atzar? Per veure si el canvi en l’ADN (timina a citosina) va ser positivament seleccionat, cal fer una ullada a la seqüència d’ADN al voltant del SNP. Si la seqüència d’ADN al voltant del rs3827760 fos similar en totes les poblacions, no tindríem cap evidència que l’SNP va tenir un efecte en l’eficàcia biològica inclusiva d’aquests organismes. Si, en canvi, una població (per exemple, la de l’est d’Àsia) va estar exposada a una pressió selectiva i el rs3827760 va contribuir al desenvolupament de l’adaptació a aquesta pressió selectiva, les seqüències d’ADN al voltant del SNP serien diferents entre les diferents poblacions. Per entendre per què això és així, vegem la figura 2.
Quan es comparen les seqüències d’ADN al voltant de rs3827760, es fa evident que la diversitat al voltant de les variants de citosina a les poblacions de l’est d’Àsia és molt més baixa que la diversitat al voltant de les variants de tiamina en poblacions d’Àfrica i Europa (les poblacions d’Amèrica no van ser estudiades). Aquests resultats suggereixen que la selecció positiva va ser responsable de l’expansió de la variant de citosina a les poblacions de l’est d’Àsia. Però va ser realment seleccionat, aquest SNP? Té això algun efecte?
No tots els canvis en les seqüències d’ADN tenen un efecte en les seqüències de proteïnes: la majoria dels SNPs catalogats a la base de dades HapMap es troben o bé en parts del genoma no codificants (és a dir, entre gens) o són sinònims, és a dir, estan localitzats en regions codificants del genoma però no produeixen canvis en la seqüència de la proteïna codificada (Figura 4).
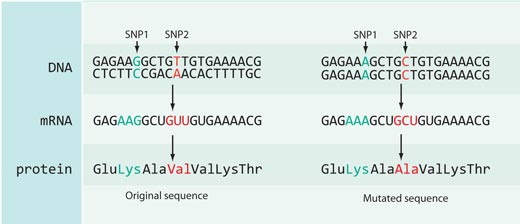
Imatge cortesia de Jarek Bryk
En el cas de rs3827760 estem de sort, perquè està localitzat en la part codificant del gen, cap al final d’un gen anomenat EDAR, el qual està involucrat en el desenvolupament dels fol·licles capil·lars, les glàndules sudorípares i les dents. A més, el canvi de timina a citosina en la seqüència d’ADN resulta en un canvi en la seqüència de la proteïna: els africans i els europeus (que tenen la variant de SNP amb timina) tenen l’aminoàcid valina en la posició 370 de la proteïna , mentre que els asiàtics de l’est i els americans (amb el nucleòtid citosina) presenten l’aminoàcid alanina. Aquesta part de la proteïna està relacionada amb interaccions amb altres proteïnes, i se sap que mutacions en aquesta zona estan relacionades amb displàsies ectodèrmiques (desenvolupament anormal de les dents, els cabells i les glàndules sudorípares) en humans i en rates (vegeu figura 5). Aquest fet suggereix que el canvi en l’aminoàcid en la posició 370 pot no tan sols canvia la seqüència de la proteïna sinó com aquesta es comporta, afectant a les característiques físiques del propi organisme.
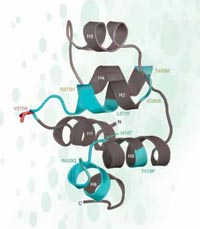
hipotètica ’d’una part de la
proteïna EDAR. Les
mutacions marcades en verd
causen displàsia ectodèrmica
en humans. L’SNP putatiu
seleccionat es presenta en
vermell
Adaptat amb permís de
Macmillan Publishers Ltd:
Nature, Sabeti et al. (2007), ©
2007
Per comprovar si el canvi en la seqüència de la proteïna afecta realment a la seva funció, hem de fixar-nos en experiments en la ruta bioquímica en la qual la proteïna EDAR participa, una sèrie de reaccions que estan involucrades en el desenvolupament de fol·licles capil·lars, glàndules sudorípares i dents. Quan aquestes reaccions es van realitzar al laboratori, es va trobar que la variant que contenia alanina (que es troba a la poblacions d’Amèrica i de l’est d’Àsia, i que està codificada per citosina en la variant de l’SNP) tornava la ruta bioquímica més activa que la variant que contenia valina (present a les poblacions africana i europea, i codificada per una timina en la variant SNP). Això està relacionat amb les comparacions entre les estructures del pèl, que van mostrar que les persones que presenten la variant alanina tenen un pèl més gruixut que les persones que presenten la variant valina. Per obtenir una demostració més directa, es van modificar genèticament ratolins per augmentar ’l’activitat de la ruta d’EDAR. Aquests ratolins presentaven un pèl visiblement més gruixut, així com glàndules salivals més grans, que els ratolins amb una activitat EDAR normal (Chunyan et al., 2008; Chang et al., 2009).
Tots aquests resultats suggereixen que les dues variants de l’SNP (que contenen bé timina bé citosina) poden afectar tant a ’l’estructura com a la funció de la proteïna EDAR, i poden portar a diferències físiques en humans: diferències en el gruix del cabell i, potencialment, en la mida de les glàndules salivals.
Les diferències en les seqüències d’ADN que podem observar avui en dia són arxius històrics d’experiments naturals, i només podem especular sobre quines de les pressions selectives a les quals les poblacions asiàtiques i americanes van ser sotmeses van ser responsables de la dispersió de ’l’al·lel citosina. Però la combinació ’d’estudis genètics, experiments de laboratori i models animals fa possible examinar les diferents hipòtesis sobre el paper funcional de les diferències genètiques entre poblacions o espècies. Utilitzant aquestes aproximacions podem descobrir les bases moleculars ’d’adaptacions passades en els nostres avantpassats i en altres organismes, destacant com ens adaptem constantment als canvis de ’l’entorn.
Glossari
Valor adaptatiu: un tret té valor adaptatiu si permet a l’individu sobreviure i reproduir-se en un entorn determinat millor que altres individus que no presenten aquest tret. De manera més formal, un tret es considera adaptatiu si augmenta ’l’eficàcia biològica inclusiva.
Eficàcia biològica inclusiva: aquest és un terme difícil de definir que prové de la biologia evolutiva i la genètica de poblacions. Descriu la descendència mitjana en una generació que està associada amb un genotip comparada amb la d’un altre genotip en la població. Per tant els genotips que produeixen més descendència tenen una eficàcia biològica adaptativa més una altra.
Genoma: quantitat total d’ADN d’una cèl.lula. Normalment en eucariotes es considera ADN nuclear, per oposició a DNA mitocondrial o plasmidi. Per a més informació, veure ‘What is a genome’ (“Què és un genoma?) a la pàgina web de la Biblioteca Nacional de Medicina dels Estats Units (US National Library of Medicine webw2).
Selecció positiva: la selecció natural és un dels mecanismes de l’evolució, que descriu les diferències de supervivència i reproducció dels individus en un entorn determinat. La selecció natural es considera positiva quan afavoreix determinats trets que ajuden a l’individuo que els presenta a sobreviure i reproduir-se millor que altres individus.
Pressió de selecció: una característica de l’entorn (per exemple temperatura, presència de paràsits, predadors o agressió per part d’altres membres de l’espècie) que imposa diferències en la supervivència i reproducció dels individus.
SNP: polimorfisme d’un únic nucleòtid, o lletra en la seqüència d’ADN que varia entre individus. Es pronuncia snip.
References
- Bryk J (2010) Natural selection at the molecular level. Science in School 14: 58-62.
- Chang SH et al. (2009) Enhanced EDAR signalling has pleiotropic effects on craniofacial and cutaneous glands. PLoS ONE 4(10): e7591. doi:10.1371/journal.pone.0007591
- Aquesta article descriu el fenotip de diverses glàndules de ratolins amb augment d’activitat en la ruta d’EDAR, i especula sobre quins trets poden haver estat seleccionats positivament en la història humana. L’article és de lliure accés a la pàgina web de la revista: www.plosone.org
- Chunyan M et al. (2008) Enhanced ectodysplasin-A receptor (EDAR) signaling alters multiple fiber characteristics to produce the East Asian hair form. Human Mutation 29(12): 1405-1411. doi: 10.1002/humu.20795
- Aquest article explica de manera detallada experiments in vitro d’EDAR y ratolins trasngènics, amb fotografies i imatges.
- Pongsophon P, Roadrangka V, Campbell A (2007) Counting Buttons: demonstrating the Hardy-Weinberg principle. Science in School 6: 30-35.
- Sabeti PC et al. (2006) Positive natural selection in the human lineage. Science312(5780): 1614-20. doi: 10.1126/science.1124309
- Aquest article presenta un excel·lent resum de diversos mètodes utilitzats en la investigació de la selecció positiva des d’una perspectiva genòmica.
- Sabeti PC et al. (2007) Genome-wide detection and characterization of positive selection in human populations. Nature 449: 913-918. doi: 10.1038/nature06250
- Aquest article es pot descarregar gratuïtament des de Science in School aquí, o per subscripció a Nature: www.nature.com/subscribe
- Aquest article descriu una de les aproximacions als estudis genòmics de selecció positiva.
- Xue Y et al (2009) Population differentiation as an indicator of recent positive selection in humans: an empirical evaluation. Genetics 183(3): 1065-77. doi:10.1534/genetics.109.107722
- Aquest article conté una discussió sobre EDAR i altres gens similanrs. Es troba disponible gratuïtament a través de PubMed Central: www.ncbi.nlm.nih.gov/pmc o utilitzant l’enllaç: http://tinyurl.com/26xte2h
Web References
- w1 –El projecte HapMap és una associació de científics i agències de finançament de Canadà, Xina, Japó, Nigèria, el Regne Unit i els EUA pel desenvolupament de recursos públics que puguin ajudar els investigadors a trobar gens associats amb malalties humanes i la seva resposta a fàrmacs. Vegeu: www.hapmap.org
- w2 – Per obtenir més informació sobre genomes i el projecte Human Genome, podeu consultar “Què és un genoma?” a la pàgina de la biblioteca Nacional de Medicina d’Estats Units (US National Library of Medicine):http://ghr.nlm.nih.gov/handbook/hgp/genome
Review
Tot i el nostre coneixement de la seqüència del genoma humà, encara es desconeix la funció exacta de grans segments de genoma, i com i per què les seqüències ’d’ADN han canviat entre poblacions. Òbviament s’ha produït evolució adaptativa en humans però és molt difícil de demostrar. Aquesta article descriu com un d’aquests canvis evolutius ha estat descobert. Experiments amb ratolins transgènics ha demostrat com un únic canvi de base en l’’ADN, que canvia la seqüència d’aminoàcids de la proteïna, condueix a una alteració de l’estructura i funció d’aquesta proteïna. Aquest canvi pot produir una variació fenotípica.
A les classes de ciències, aquest article es pot utilitzar per il·lustrar els temes de l’ús i degeneració de codons, estructura i funció de proteïnes, i genètica de poblacions. Pot ser utilitzat també com a lectura complementària en els temes de variació en poblacions humanes o com a punt de partida per investigar l’Institut Sanger i el Projecte Human Genome.
Els alumnes poden discutir sobre avantatges evolutius, fent referència a la variació descrita en aquest article. Això podria portat a una discussió sobre selecció, genètica de poblacions i equilibri de Hardy-Weinberg. Per completar la discussió, hi ha una excel.lent activitat en el número 6 de Science in School (Pongsophon et al., 2007).
Algunes preguntes de comprensió poden ser:
- En les teves pròpies paraules i donant un exemple que no es trobi en el text, descriu què és un SNP.
- Explica la importància dels SNPs.
- Per quin aminoàcid codifica el triplet GTT?
- Descriu els canvis que es van trobar en els ratolins genèticament modificats en la ruta d’EDAR, i suggereix maneres per quantificar aquets canvis.
Shelley Goodman, Regne Unit





