Evolution humaine: tester l’hypothèse de la base moléculaire Understand article
Traduit par Maurice A. Casimir. Dans un second et dernier article, Jarek Bryk décrit la manière dont les scientifiques plongent au cœur de nos gènes – pour tester la base moléculaire d’une adaptation de l’évolution chez les êtres humains.

autorisation de Henrik500 /
iStockphoto
L’ADN de chaque organisme détient des informations sur son évolution, récente et ancienne. L’étude des modèles et modifications de la séquence d’ADN – par comparaison des séquences entre différents individus ou espèces – permet de découvrir ce qu’il leur est arrivé. On peut connaître les gènes ou fragments du génome dont il est probable qu’ils ont fourni un avantage évolutionniste à ces individus ou espèces chez qui ils sont présents, en leur permettant de mieux survivre et se reproduire (voir glossaire pour tous les termes en gras).
J’ai décrit dans un article précédent (Bryk, 2010) quelques exemples de telles modifications génétiques bénéfiques chez les êtres humains et autres organismes. Montrer quelles modifications génétiques peuvent avoir été bénéfiques est difficile – particulièrement chez les êtres humains – mais trouver le mécanisme selon lequel ces modifications ont pu améliorer la survie et la reproduction des organismes représente un défi encore plus important.
Je présente dans l’article l’une des approches que les scientifiques ont utilisées tout d’abord pour identifier les régions du génome qui auraient pu nous aider à survivre et à nous reproduire, puis pour tester la manière dont ces régions ont pu créer un avantage chez nos ancêtres.
L’une des façons simples d’identifier les régions potentiellement bénéfiques de notre génome est de comparer les séquences d’ADN de nombreux individus issus de populations différentes. Selon un scénario très simple, si l’une de ces populations avait été l’objet d’une pression sélective (par exemple présence d’un fort rayonnement UV dans une région ensoleillée), absente chez d’autres populations, la séquence d’ADN responsable d’une adaptation appropriée (par exemple un peau de couleur foncée) devrait être différente.
Adapté avec la permission de Macmillan Publishers Ltd: Nature, Sabeti et al. (2007), © 2007
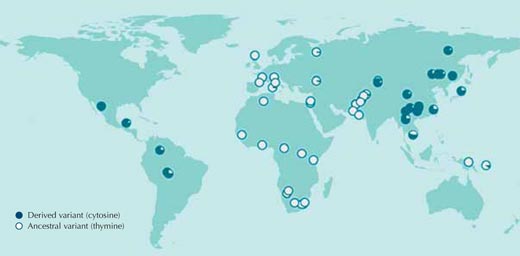
Dans la grande majorité des cas, toutefois, nous ne savons pas à quelles pressions sélectives les populations ont été exposées dans le passé, ou quelles séquences génétiques sont responsables des adaptations. Pour commencer, par conséquent, il faut comparer les séquences d’ADN entre populations humaines sans émettre aucune hypothèse sur ce que nous pourrions trouver. La Figure 1 montre une telle comparaison pour un nucléotide isolé du génome humain.
Lorsque des individus présentent des nucléotides differents pour une position particulière de la séquence d’ADN, on parle de polymorphisme nucléotidique simple (SNP, à prononcer ‘snip’) ; trois millions de telles variantes du génome humain sont cataloguées dans la base de données HapMapw1 ouverte au public. Le SNP de la Figure 1, rs3827760, se trouve chez deux variantes ou allèles: à cet endroit de la séquence, on peut trouver l’une de deux bases – soit la thymine (T) soit la cytosine (C).
Chaque cercle de la figure représente une population unique et montre la fréquence chez cette population des deux allèles possibles.
L’allèle contenant la thymine est présent chez tous les Africains et chez la plupart des Européens, mais est presque complètement absent en Asie orientale et aux Amériques, où la cytosine prévaut dans cette position de la séquence (Sabeti et al., 2007, 2006; Xue et al., 2009).
Si nous avions fait cette comparaison pour les autres trois milllions de SNP du HapMap nous aurions constaté que la distribution des variantes de rs3827760 parmi les populations humaines est très inhabituelle. rs3827760 mérite en définitive qu’on y regarde de plus près, même si la distribution ne nous apprend rien au sujet des bénéfices potentiels des variantes (leur valeur d’adaptation), ni même s’ils sont adaptatifs ou non. Tout ce que nous savons à ce jour est que, pour une certaine raison, la thymine d’origine qui était présente à cet endroit chez les populations humaines de nos ancêtres africains s’est transformée en cytosine, et que ce changement s’est répandu chez les Asiatiques de l’Est et chez les Américains. Même l’estimation de l’époque de ce changement est très imprécise : à un moment donné entre il y a 1.000 et 70.000 ans, tous les individus des populations de l’Asie de l’Est ont acquis la variante cytosine.
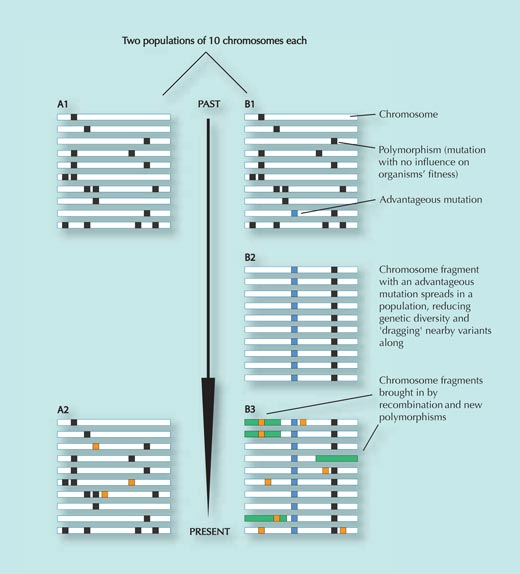
La population A ne fait pas l’objet d’une pression sélective et reste relativement inchangée dans le temps, sauf par acquisition de quelques modifications génétiques aléatoires n’influençant pas ses aptitudes (carrés orange dans le panneau 2 ; les variantes affectant les aptitudes sont retirées de la population) – comparer les panneaux 1 et 2.
Si toutefois la population B se déplace vers un nouvel environnement, elle fait face à de nouvelles pressions sélectives. Dans cet environnement nouveau, une modification génétique particulière (le carré bleu sur le panneau B1) fournit un avantage aux individus qui en sont porteurs et se répand rapidement parmi la population (les individus porteurs ont une descendance plus nombreuse). Les variantes génétiques proches du SNP sélectionné y restent attachées (plus deux variantes sont proches, plus la chance qu’elles soient séparées durant la recombinaison, lorsque les parties d’ADN sont échangées entre les chromosomes maternel et paternel, est faible – voir Figure 3).
Après un certain temps, cependant, de nouvelles modifications génétiques et la recombinaison introduisent de nouvelles variantes (les rectangles verts et les carrés orange dans le panneau B3). Plus il s’est écoulé de temps depuis la dissémination de la variante sélectionnée d’origine, plus il est difficile de la détecter, car le cheminement de la diversité réduite (B2) sera masqué à la longue (B3)
Image reproduite avec l’aimable autorisation de Jarek Bryk
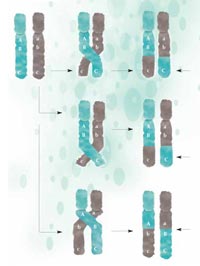
recombinaison, des parties
de l’ADN sont échangées
entre les chromosomes
maternels (verts) et paternels
(gris) et transférés à la
génération suivante en une
configuration nouvelle. Plus
deux régions sont proches,
moins il est probable qu’elles
se séparent durant la
recombinaison : A et B sont
plus proches l’un de l’autre
que B et C, et seront moins
probablement séparés.
Cliquer sur l’image pour
l’agrandir
Image reproduite avec
l’aimable autorisation de Nicola
Graf
Comment alors decider si ce modèle s’est développé du fait d’une sélection positive (la cytosine conférait un avantage aux populations d’Asie de l’Est et aux Américains) ou s’il est simplement dû au hasard ? Pour examiner si la modification d’ADN (de la thymine à la cytosine) a été due à une sélection positive, il faut considérer la séquence d’ADN autour du SNP. Si la séquence d’ADN entourant rs3827760 est similaire chez toutes les populations, nous n’aurons aucune preuve de ce que le SNP a eu un effet sur les aptitudes. Si toutefois une population (d’Asie de l’Est, par exemple) a été exposée à une pression sélective et si rs3827760 a contribué au développement d’une adaptation à cette pression sélective, les séquences d’ADN autour du SNP différeraient entre les populations. Pour comprendre pourquoi il en est ainsi, voir Figure 2.
Lorsque l’on compare les séquences d’ADN autour de rs3827760, il devient évident que la diversité autour des variantes de cytosine est en fait bien inférieure chez les populations d’Asie de l’Est à la diversité autour des variantes de thymine chez les populations d’Afrique et d’Europe (l’Amérique n’a pas été testée). Ceci laisse penser que la sélection positive a été responsable de la dissémination de la variante de cytosine chez les populations d’Asie de l’Est. Mais ce SNP a-t-il réellement été sélectionné – a-t-l en fait une action quelconque?
Les modifications à la séquence d’ADN n’ont pas toutes un effet sur les séquences de protéines : la plupart des SNP catalogués dans la base de données HapMap sont soit localisés dans des parties non codantes du génome (c.à.d. entre des gènes) ou sont synonymes – c’est-à-dire qu’ils sont localisés dans la partie codante du génome mais sans amener de modification dans la séquence de protéine encodée (voir Figure 4).
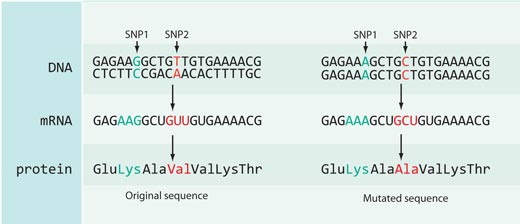
Image reproduite avec l’aimable autorisation de Jarek Bryk
Dans le cas de rs3827760, nous avons de la chance, car il est situé dans la partie codante d’un gène – vers l’extrémité d’un gène appelé EDAR, qui est impliqué dans le développement des follicules pileuses, des glandes sudoripares et des dents. En outre, la modification dans la séquence d’ADN de thymine en cytosine a pour résultat de modifier la séquence de protéine : les Africains et les Européens (porteurs de la variante SNP à thymine) ont l’aminoacide valine en position 370 sur la protéine, alors que les Asiatiques de l’Est et les Américains (porteurs du nucléotide cytosine) ont l’aminoacide alanine. Cette partie de la protéine est impliquée dans des interactions avec d’autres protéines, et dans des mutations dont l’on sait qu’elles causent la dysplasie ectodermique – un développement anormal des dents, des poils et des glandes sudoripares – chez les humains et les souris (voir Figure 5). Ce fait laisse fortement penser qu’une modification d’aminoacide en position 370 pourrait changer non seulement la séquence de la protéine mais également son comportement, en affectant les caractéristiques physiques de l’organisme lui-même.
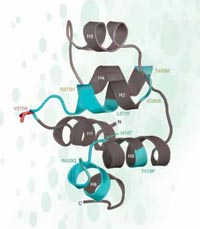
hypothétique d’une partie de
la protéine EDAR. Les
mutations marquées en vert
causent la dysplasie
ectodermique chez les
humains. Le SNP sélectionné
généralement reconnu est
marqué en rouge
Adapté avec la permission de
Macmillan Publishers Ltd:
Nature, Sabeti et al. (2007), ©
2007
Pour voir si la modification dans la séquence de protéine affecte effectivement sa fonction, nous nous sommes tournés vers des expériences sur les chaînes de réactions biochimiques auxquelles la protéine EDAR prend part : une série de réactions impliquées dans le développement des follicules pileuses, des glandes sudoripares et des dents. Lorsque ces réactions furent réalisées au laboratoire, la variante alanine de la protéine (présente chez les Asiatiques de l’Est et les Américains, encodée par la variante SNP de la cytosine) s’avéra rendre la réaction plus active que dans le cas de la variante valine (présente chez les Africains et les Européens, encodée par la variante SNP de la thymine). Cela s’accorde avec des comparaisons de la structure pileuse, qui montre que les personnes avec la variante alanine ont des cheveux plus épais que les personnes avec la variante valine. Pour une démonstration plus directe, des souris furent génétiquement modifiées pour accroître l’activité de la chaîne de réactions EDAR. Ces souris avaient une fourrure visiblement plus dense avec des poils plus épais, ainsi que des glandes salivaires plus grandes que celles montrant une activité EDAR normale (Chunyan et al., 2008;Chang et al., 2009).
Ces découvertes prises ensemble laissent penser que les deux variantes SNP (contenant soit de la thymine, soit de la cytosine) pourraient affecter à la fois la structure et la fonction de la protéine EDAR, et pourraient conduire à des différences physiques chez les humains : des différences dans l’épaisseur des cheveux et, potentiellement, la taille des glandes salivaires.
Les différences observées aujourd’hui dans les séquences d’ADN sont des rappels historiques d’expériences de la nature, et nous pouvons uniquement spéculer sur les pressions sélectives auxquelles furent exposées les populations asiatiques et américaines et qui encouragèrent la dissémination de l’allèle cytosine. Mais la combinaison des études génomiques, des expériences de laboratoire et des modèles animaux rend possible les tests sur des hypothèses concernant les rôles fonctionnels des différences génétiques entre populations ou espèces. En utilisant cette approche, nous pourrons révéler la base moléculaire des adaptations passées chez nos ancêtres et chez d’autres organismes, en mettant en lumière la manière dont nous nous adaptons à un environnement changeant en permanence.
Glossaire
Valeur d’adaptation: un trait a une valeur d’adaptation s’il permet à un individu de mieux survivre et se reproduire dans un environnement donné que des individus ne possédant pas ce trait. De façon plus formelle, un trait est considéré comme adaptatif s’il augmente les aptitudes.
Allèle: une variante d’un gène.
Aptitude: Un terme formellement difficile à définir à partir de la biologie évolutionniste et de la génétique des populations ; il a trait au nombre moyen de descendants sur une génération associés à un génotype par comparaison à un autre génotype d’une population. Les génotypes produisant plus de descendants disposent d’une meilleure aptitude.
Génome: L’ADN total d’un organisme. Habituellement compris comme ADN total chez les eucharyotes, par opposition à l’ADN incluant mitochondries ou plastides. Pour en savoir plus, voir ‘Qu’est-ce qu’un génome’ sur le site Web de la « US National Library of Medicinew2».
Sélection positive: la selection naturelle est l’un des mécanismes de l’évolution; il décrit la survie et la reproduction différenciées des individus dans un environnement donné. La sélection naturelle est appelée ‘positive’ lorsqu’elle privilégie certains traits aidant les individus à survivre et se reproduire mieux que d’autres.
Pression sélective: une caractéristique de l’environnement (comme température ; présence de parasites ; prédation ou agression par des membres des mêmes espèces) qui impose un survie et une reproduction différenciée des individus.
SNP: un polymorphisme nucléotidique simple, ou une seule lettre dans la séquence d’ADN différant entre individus. Prononcer ‘snip’.
References
- Bryk J (2010) La sélection naturelle vue au niveau moléculaire. Science in School14: 58-62.
- Chang SH et al. (2009) Enhanced EDAR signalling has pleiotropic effects on craniofacial and cutaneous glands. PLoS ONE 4(10): e7591. doi:10.1371/journal.pone.0007591
- Cet article décrit le phénotype de diverses glandes de souris avec un signal EDAR amélioré , et spécule sur les traits qui pourraient avoir connu une sélection positive lors de l’histoire de l’humanité. L’article est disponible gratuitement sur le site Web du journal: www.plosone.org
- Chunyan M et al. (2008) Enhanced ectodysplasin-A receptor (EDAR) signaling alters multiple fiber characteristics to produce the East Asian hair form. Human Mutation 29(12): 1405-1411. doi: 10.1002/humu.20795
- Cet article détaille les etudes ‘in vitro’ de l’EDAR et des souris transgéniques, avec de très belles images et photos.
- Pongsophon P, Roadrangka V, Campbell A (2007) Counting Buttons: demonstrating the Hardy-Weinberg principle. Science in School 6: 30-35.
- Sabeti PC et al. (2006) Positive natural selection in the human lineage. Science312(5780): 1614-20. doi: 10.1126/science.1124309
- Il s’agit d’une excellente vue d’ensemble des diverses méthodes utilisées pour rechercher la sélection positive du point de vue de la génomique.
- Sabeti PC et al. (2007) Genome-wide detection and characterization of positive selection in human populations. Nature 449: 913-918. doi: 10.1038/nature06250
- Téléchargez gratuitement l’article à partir de Science in School ici, ou abonnez-vous à Nature dès aujourd’hui: www.nature.com/subscribe
- Cet article décrit l’une des approches des recherches au niveau du génome sur la sélection positive.
- Xue Y et al (2009) Population differentiation as an indicator of recent positive selection in humans: an empirical evaluation. Genetics 183(3): 1065-77. doi:10.1534/genetics.109.107722
- Cet article contient une discussion sur l’EDAR et autres gènes similaires. Il est disponible gratuitement via PubMed Central: www.ncbi.nlm.nih.gov/pmc ou en utilisant le lien direct: http://tinyurl.com/26xte2h
Web References
- w1 – Le projet HapMap s’appuie sur un partenariat de scientifiques et d’agences de financement du Canada, du Japon, du Nigéria, du Royaume Uni et des Etats Unis pour développer une source publique d’aide aux chercheurs pour la découverte de gènes associés aux maladies humaines et pour leur traitement à partir de médicaments. Voir: www.hapmap.org
- w2 – Pour en savoir davantage sur les génomes et le Projet de Génome Humain, voir ‘Qu’est-ce qu’un génome’ sur le site Web de la « US National Library of Medicine: http://ghr.nlm.nih.gov/handbook/hgp/genome
Review
En dépit de tout ce que nous savons sur la séquence du génome humain, la fonction précise d’énormes segments de celui-ci et la manière dont les séquences d’ADN ont changé dans des populations ainsi que la raison de ce changement restent largement à découvrir. Les adaptations évolutionnistes chez les humains se sont vraiment produites, mais cela reste très difficile à démontrer. Cet article décrit la manière dont une telle modification a été identifiée. Des expériences avec des souris génétiquement modifiées ont montré comment une simple modification de base dans l’ADN, modifiant la séquence des aminoacides de la protéine, conduit à une altération de la structure et de la fonction d’une protéine. Ceci peut conduire à une variation du phénotype.
Dans les leçons de science, on pourrait utiliser l’article lorsque l’on traite des thèmes de la manipulation et de la dégénérescence du codon ainsi que de la génétique de la population. On pourrait également l’utiliser comme lecture de base sur les variations chez la population humaine ou comme point de départ pour des recherches sur le Sanger Institute et le Projet de Génome Humain.
Les élèves pourraient discuter de l’avantage évolutionniste, en se référant à la variation particulière décrite dans l’article. Ceci pourrait entraîner une discussion sur la sélection, la génétique de la population et l’équilibre de Hardy-Weinberg. Pour compléter cette discussion, il existe une excellente présentation dans le numéro 6 de Science in School (Pongsophon et al., 2007).
Des questions adéquates portant sur la comprehension:
- Décrire ce qu’est un SNP de façon originale et en donnant un exemple non dans le texte.
- Expliquer ce que sont les SNP.
- Quel aminoacide encode dans le nucleotide le triplet GTT?
- Décrire les modifications découvertes dans les souris soumises à une chaîne de réactions sur un EDAR génétiquement modifié, et suggérer des manières de pouvoir quantifier les modifications observées.
Shelley Goodman, Royaume Uni





