Evolução humana: uma análise da base molecular Understand article
Traduzido por: Artur Melo. Neste segundo de dois artigos, Jarek Bryk descreve como os cientistas avançam no conhecimento sobre os nossos genes – na análise da base molecular de uma adaptação evolutiva em humanos

iStockphoto
O ADN de todos os organismos guarda informações sobre a sua história recente e passada. Estudando os padrões e alterações na sequência do ADN – pela comparação das sequências entre diferentes individuos e espécies – podemos descobrir o que lhes aconteceu. Podemos saber quais os genes ou fragmentos do genoma que poderão ter atribuido vantagem aos indivíduos e espécies que os transportaram, permitindo-lhes melhor sobrevivencia e reprodução (ver o glossário para todos os termos a bold).
Num artigo anterior (Bryk, 2010), abordei alguns exemplos destas alterações genéticas benéficas, em humanos e outros organismos. A demonstração de quais as alterações genéticas que poderão ter sido benéficas é difícil – especialmente em humanos – mas um desafio ainda maior é demonstrar o mecanismo pelo qual estas alterações poderão ter melhorado a sobrevivência e reprodução dos organismos.
Neste artigo, apresento uma das abordagens que os cientistas podem usar para identificar regiões do nosso genoma que poderão ter ajudado a sobrevivência e a reprodução, e em seguida testar como estas regiões terão fornecido aos nossos antepassados vantagens.
Uma das formas pela qual as regiões potencialmente benéficas do nosso genoma podem ser identificadas é pela simples comparação das sequências de ADN de muitos indivíduos de diferentes populações. Num cenário simplificado, se uma destas populações foi sujeita a uma pressão selectiva (por exemplo, radiação UV elevada numa região ensolarada) ausente nas outras populações, a sequência de ADN responsável por uma adaptação adequada (por exemplo, uma cor de pele mais escura) deverá ser diferente.
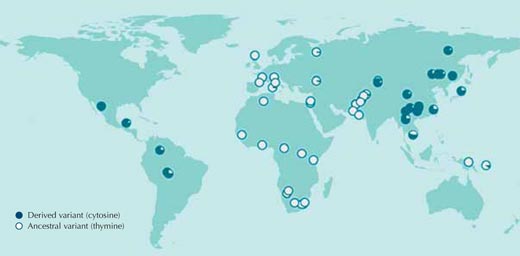
Adaptado com permissão de Macmillan Publishers Ltd: Nature, Sabeti et al. (2007), © 2007
Na vasta maioria dos casos, no entanto, nós não conhecemos quais as pressões selectivas a que estiveram expostas as populações no passado, ou que sequências genéticas são responsáveis pelas adaptações. Comecemos, por isso, pela comparação de sequências de ADN entre populações humanas, sem quaisquer previsões do que podemos encontrar. A figura 1 mostra uma dessas comparações num nucleótido do genoma humano.
Quando os indivíduos apresentam nucleótidos diferentes numa determinada posição da sequência de ADN, ela é designada por single nucleotide polymorphism(SNP, que se pronuncia ‘snip’); três milhões destas variações no genoma humano estão catalogadas na base de dados HapMapw1 acessível ao público. O SNP da figura 1, rs3827760, ocorre em duas variantes ou alelos: neste ponto da sequência, pode encontrar-se uma de duas bases – ou timina (T) ou citosina (C).
Cada círculo na figura representa uma população, e inclui a frequência naquela população dos dois alelos possíveis.
O alelo com timina está presente nas amostras de todos os africanos e da maioria dos europeus, mas está quase completamente ausente na Ásia Oriental e nas Américas, onde a citosina prevalece nessa posição da sequência (Sabeti et al., 2007, 2006; Xue et al., 2009).
Se fizessemos esta comparação com todos os outros três milhões de SNPs registados no HapMap, veríamos que a distribuição das variantes de rs3827760 nas populações humanos é bastante invulgar. Assim, o rs3827760 merece, sem dúvida, uma observação mais detalhada, embora a distribuição não nos informe sobre o potencial benefício das variantes (o seu valor adaptativo), ou mesmo se têm algum valor adaptativo. Tudo o que sabemos até agora é que, por alguma razão, a timina que inicialmente estava presente nesta posição nas populações humanas ancestrais em África, mudou para citosina, e que esta alteração se espalhou pelos asiáticos orientais e americanos. Mesmo as estimativas do momento em que a alteração ocorreu, são muito imprecisas: algures entre 1000 e 70 000 anos atrás, todos os indivíduos das populações da Ásia Oriental tinham a variante de citosina.
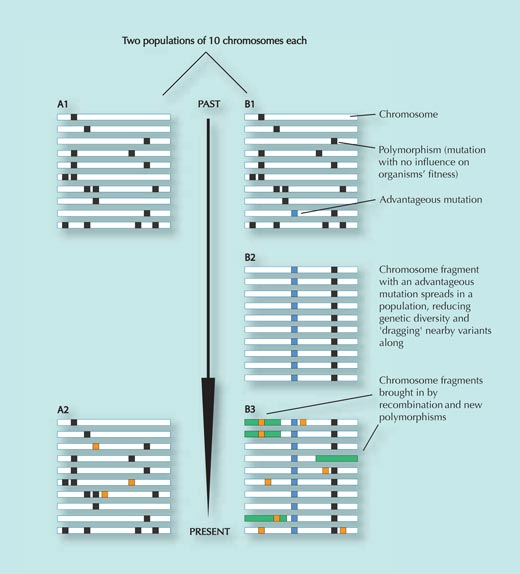
A população A não esteve sujeita a pressão selectiva positiva e permanece relativamente inalterada ao longo do tempo, apesar de adquirir algumas alterações genéticas aleatórias que não influenciam a aptidão (quadrados laranja no painel A2; as variantes que reduzem a aptidão são removidas da população) – compare os painéis A1 e A2.
Contudo, a população B desloca-se para um ambiente novo, onde enfrenta novas pressões selectivas. Nesse novo ambiente, uma determinada alteração genética (o quadrado azul no painel B1) atribui uma vantagem aos indivíduos que a transportam e rapidamente se espalha pela população (os indivíduos que a têm produzem mais descendentes). As variantes genéticas próximas do SNP seleccionado são arrastadas com ela (quanto mais próximas estiverem as duas variantes, menor a probabilidade de se separarem durante a recombinação, em que fragmentos de ADN são trocados entre os cromossomas materno e paterno – ver a figura 3).
O resultado deste rápido alastramento de uma sequência de ADN pela população é a redução na diversidade genética nessa região; a maior parte dos indivíduos terá o SNP vantajoso, juntamente com as variantes genéticas que ficam próximas (compare os painéis B1 e B2). Este processo ocorre rapidamente.
Após algum tempo, no entanto, novas alterações genéticas e fenómenos de recombinação introduzem novas variantes (rectângulos verdes e quadrados laranja no painel B3). Quanto mais tempo decorrer desde o alastramento da variante seleccionada original, mais difícil é de detectar, porque o padrão de diversidade reduzida (B2) ficará eventualmente escondido (B3)
Imagem cortesia de Jarek Bryk
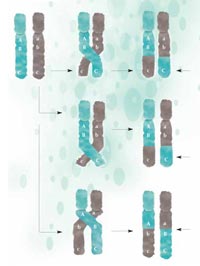
recombinação, fragmentos do
ADN são trocados entre os
cromossomas materno
(verde) e paterno (cinzento) e
transmitidos à geração
seguinte numa nova
configuração. Quanto mais
próximas duas regiões
estiverem, menor a
probabilidade de se
separarem durante a
recombinação: A e B estão
mais próximas que B e C,
sendo, portanto, menos
susceptíveis de serem
separadas. Clique para
ampliar a imagem
Imagem cortesia de Nicola Graf
Como podemos, então, definir se este padrão surgiu devido a selecção positiva (a citosina conferia vantagem na Ásia Oriental e nas Américas) ou simplesmente por acaso? Para determinar se a alteração no ADN (timina para citosina) foi seleccionada positivamente, verificamos a sequência de ADN próxima do SNP. Se a sequência de ADN próxima do rs3827760 for semelhante em todas as populações, não podemos afirmar que o SNP teve efeito na aptidão do organismo. Se, no entanto, uma população (asiática oriental, por exemplo) esteve exposta a uma pressão selectiva e o rs3827760 contribuiu para o desenvolvimento de adaptação a essa pressão selectiva, as sequências de ADN próximas do SNP seriam diferentes nas várias populações. Para perceber porque é assim, veja a figura 2.
Quando as sequências de ADN próximas do rs3827760 são comparadas, torna-se evidente que a diversidade das variantes de citosina nas populações da Ásia Oriental é, de facto, muito menor que a diversidade das variantes de timina encontradas nas populações de África e da Europa (as americanas não foram analisadas). Isto sugere que a selecção positiva foi responsável pelo alastramento da variante citosina nas populações do leste asiático. Mas foi este SNP realmente seleccionado – tem de facto algum efeito?
Nem todas as alterações na sequência do ADN têm efeito na sequência das proteínas: a maioria dos SNPs catalogados na base de dados HapMap localizam-se em zonas não codificantes do genoma (isto é, entre genes) ou são silenciosos – isto é, localizam-se em zonas codificantes do genoma mas não provocam alteração na sequência da proteína codificada (ver Figura 4).
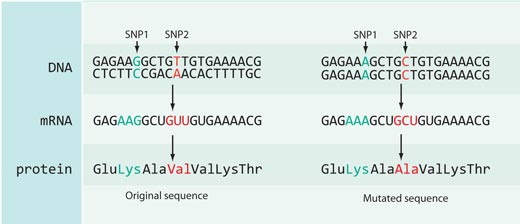
Imagem cortesia de Jarek Bryk
No caso do rs3827760 temos sorte, porque está localizado na região codificante do gene – na extremidade de um gene chamado EDAR, que está envolvido no desenvolvimento de folículos capilares, de glândulas sudoríparas e dos dentes. Além disso, a alteração de timina para citosina na sequência do ADN provoca a alteração da sequência de aminoácidos na proteína: os africanos e os europeus (que possuem a variante de SNP com timina) têm o aminoácido valina na posição 370 da proteína, enquanto os leste asiáticos e os americanos (com o nucleótido citosina) têm o aminoácido alanina. Esta região da proteína está envolvida em interacções com outras proteínas, e sabe-se que mutações neste ponto causam displasias ectodérmicas – desenvolvimento anormal de dentes, cabelo e glândulas sudoríparas – em humanos e ratos (ver Figura 5). Este facto sugere que uma alteração do aminoácido na posição 370 pode não apenas alterar a sequência da proteína mas, também, o comportamento da proteína, influenciando as características físicas do próprio organismo.
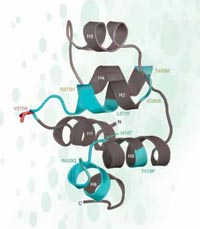
de parte da proteína EDAR.
As mutações assinaladas a
verde causam displasia
ectodérmica em humanos. O
SNP supostamente
seleccionado está
representado a vermelho
Adaptado com permissão de
Macmillan Publishers Ltd:
Nature, Sabeti et al. (2007), ©
2007
Para verificar se a alteração na sequência da proteína realmente afecta a sua função, realizamos experiências no âmbito da via bioquímica em que a proteína EDAR participa: uma série de reacções envolvidas no desenvolvimento de folículos pilosos, glândulas sudoríparas e dentes. Quando estas reacções foram realizadas no laboratório, descobriu-se que a variante de alanina da proteína (encontrada em leste asiáticos e americanos, codificada pela variante SNP de citosina) tornava a via mais activa do que a variante de valina (encontrada em africanos e europeus, codificada pela variante SNP de timina). Isto é compatível com as comparações da estrutura capilar, que mostram que as pessoas com a variante de alanina possuem cabelos mais espessos que as pessoas com a variante de valina. Para uma demonstração mais directa, foram geneticamente modificados ratos para aumentar a actividade da via da EDAR. Estes ratos apresentavam pelagem visivelmente mais densa com pêlos mais espessos, assim como glândulas salivares maiores, do que os ratos com actividade da EDAR normal (Chunyan et al., 2008; Chang et al., 2009).
Consideradas em conjunto, estas descobertas sugerem que as duas variantes de SNP (com timina ou citosina) podem influenciar tanto a estrutura como a função da proteína EDAR, e podem originar diferenças físicas em humanos: diferenças na espessura do cabelo e, potencialmente, o tamanho das glândulas salivares.
As diferenças nas sequências de ADN que observamos hoje são registos históricos de ensaios naturais, e apenas podemos especular sobre as pressões selectivas a que as populações asiáticas e americanas estiveram expostas, as quais encorajaram o alastramento do alelo de citosina. Mas a combinação de estudos genómicos, experiências laboratoriais e modelos animais torna possível testar hipóteses sobre os papéis funcionais das diferenças genéticas entre populações ou espécies. Usando estas abordagens, podemos revelar a base molecular de adaptações passadas, nos nossos antepassados e outros organismos, evidenciando como nos adaptamos a um ambiente em constante alteração.
Glossário
Valor adaptativo: um carácter tem valor adaptativo se permite a um indivíduo sobreviver e reproduzir-se melhor, num determinado ambiente, do que os indivíduos que não apresentam esse carácter. Mais formalmente, um carácter é considerado como adaptativo se aumenta a aptidão.
Alelo: uma variante de um gene.
Aptidão: um termo de difícil definição em biologia evolutiva e genética de populações; refere-se ao número médio de descendentes, durante uma geração, associado a um genótipo comparado com outro genótipo numa população. Assim, genótipos que produzem mais descendência têm maior aptidão.
Genoma: o ADN total de um organismo. Normalmente considerado em eucariotas como o ADN nuclear total, em oposição à inclusão do ADN mitocondrial ou dos plastos. Para mais informações, ver “What is a genome” no website da “National Library of Medicine” dos EUAw2.
Selecção positiva: a selecção natural é um dos mecanismos de evolução; refere-se à sobrevivência e reprodução diferencial dos indivíduos num determinada ambiente. A selecção natural é chamada ‘positiva’ quando favorece determinados caracteres que ajudam os indivíduos a sobreviver e reproduzir-se melhor que os outros.
Pressão selectiva: um factor do ambiente (por exemplo, temperatura, presença de parasitas, predação ou agressão de elementos da mesma espécie) que impõe a sobrevivência e reprodução diferencial dos indivíduos.
SNP: polimorfismo de um único nucleótido, ou uma letra na sequência do ADN que é diferente entre indivíduos. Pronuncia-se ‘snip’.
References
- Bryk J (2010) Selecção natural a nível molecular. Science in School 14: 58-62.
- Chang SH et al. (2009) Enhanced EDAR signalling has pleiotropic effects on craniofacial and cutaneous glands. PLoS ONE 4(10): e7591. doi: 10.1371/journal.pone.0007591
- Este artigo descreve o fenótipo de várias glândulas em ratos com sinalização reforçada EDAR, e especula sobre quais os caracteres que poderiam ter sido positivamente seleccionados na história humana. O artigo está gratuitamente disponível no website da revista: www.plosone.org
- Chunyan M et al. (2008) Enhanced ectodysplasin-A receptor (EDAR) signaling alters multiple fiber characteristics to produce the East Asian hair form. Human Mutation 29(12): 1405-1411. doi: 10.1002/humu.20795
- Este artigo pormenoriza estudos in vitro da EDAR e ratos transgénicos, com imagens e fotografias muito boas.
- Pongsophon P, Roadrangka V, Campbell A (2007) Counting Buttons: demonstrating the Hardy-Weinberg principle. Science in School 6: 30-35.
- Sabeti PC et al. (2006) Positive natural selection in the human lineage. Science 312(5780): 1614-20. doi: 10.1126/science.1124309
- Este é um excelente resumo de vários métodos usados na investigação da selecção positiva numa perspectiva genómica.
- Sabeti PC et al. (2007) Genome-wide detection and characterization of positive selection in human populations. Nature 449: 913-918. doi: 10.1038/nature06250
- Descarregue o artigo gratuitamente aqui, ou subscreva a Nature agora: www.nature.com/subscribe
- Este documento descreve uma das abordagens à investigação genómica da selecção positiva.
- Xue Y et al (2009) Population differentiation as an indicator of recent positive selection in humans: an empirical evaluation. Genetics 183(3): 1065-77. doi: 10.1534/genetics.109.107722
- Este documento contém argumentação sobre o EDAR e outros genes semelhantes. É de acesso grátis através da PubMed Central: www.ncbi.nlm.nih.gov/pmc ou através do link directo: http://tinyurl.com/26xte2h
Web References
- w1 – O projecto HapMap é uma parceria de cientistas e Fundações do Canadá, China, Japão, Nigéria, Reino Unido e EUA para o desenvolvimento de recursos públicos que ajudem os investigadores a descobrir genes associados a doenças humanas e a reacção a medicamentos. Ver: www.hapmap.org
- w2 – Para mais informação sobre genomas e o Projecto Genoma Humano, ver ‘What is a genome’ no website da ‘National Library of Medicine’ dos EUA:http://ghr.nlm.nih.gov/handbook/hgp/genome
Review
Apesar de todo o conhecimento da sequência do genoma humano, a função exacta de grandes segmentos que o constituem, e de como e porquê as sequências de ADN têm mudado nas populações, permanece na sua maior parte por descobrir. A adaptação evolutiva em humanos aconteceu obviamente, mas é bastante difícil demonstrá-lo. Este artigo descreve como uma dessas alterações foi identificada. Experiências com ratos geneticamente modificados demonstraram como a alteração numa única base do ADN, que modifica a sequência de aminoácidos da proteína, conduz a uma alteração na estrutura e função de uma proteína. O que pode resultar numa variação fenotípica.
Em aulas de ciências, o artigo pode ser usado na apresentação de conceitos sobre codões e degenerescência; estrutura e função das proteínas; e genética de populações. Pode também ser usado como leitura de apoio sobre diversidade em populações humanas ou como início de estudo do ‘Sanger Institute’ e do ‘Human Genome Mapping Project’.
Os alunos podem debater a vantagem evolutiva, com referência especial à variação descrita neste artigo. Isto poderá levar a um debate sobre selecção, genética de populações e equilíbrio de Hardy-Weinberg. Para complementar este debate, existe uma actividade excelente na Science in School número 6 (Pongsophon et al., 2007).
Algumas questões de compreensão adequadas:
- Por palavras suas e através de um exemplo que não se encontre no texto, descreva o que é um SNP.
- Explique a importância dos SNPs.
- Qual o aminoácido que o tripleto de nucleótidos GTT codifica?
- Descreva as alterações que foram descobertas em ratos com uma via EDAR geneticamente modificada, e sugira formas de quantificar as alterações observadas.
Shelley Goodman, Reino Unido





