Svante Pääbo: un arqueólogo del genoma Understand article
Traducido por José Luis Cebollada. Svante Pääbo, investigador en genética evolutiva explica a Eleanor Hayes como se explora el genoma para comprender la evolución humana.

Training Centre del European
Molecular Biology Laboratory
en Heidelberg, Alemania.
¿Puedes ver el parecido con
la doble hélice de ADN? Haga
clic sobre la imagen para
ampliarla
Imagen cortesía de EMBL
Photolab
Siempre había imaginado a los arqueólogos arrodillados en un lugar polvoriento, excavando huesos, joyas antiguas o restos de vasijas. Cuando conocí a Svante Pääbo, un ‘arqueólogo del genoma’ ni estaba arrodillado ni trabajaba al aire libre.
Por el contrario, nos encontramos –como no podía ser de otra manera – en el flamante edificio del Advanced Training Centre del European Molecular Biology Laboratory en Heidelberg, Alemania. Su arquitectura refleja la molécula de ADN, los edificios tienen dos pasillos helicoidales con despachos y laboratorios conectados por puentes de cristal que representan los puentes de hidrógenow1.
¿Qué relación hay entre la arqueología y el ADN? “Mis colegas y yo hacemos en cierta forma lo mismo que los arqueólogos”, explica el profesor Pääbo. “No excavamos en una fosa antigua sino en el genoma. Estudiamos secuencias de nuestro ADN buscando vestigios de nuestra historia, por ejemplo para averiguar de dónde procedemos y cómo hemos interaccionado con otros humanos.”
Le pregunto ¿Cómo llegó a este campo de investigación? Habla de su temprana fascinación por Egipto, acrecentada por unas vacaciones con su madre en ese país, que le llevó a estudiar Egiptología en la universidad de Uppsala, Suecia. Sin embargo, una vez allí, sus sueños románticos se desvanecieron: “Estudiábamos más sobre las palabras en egipcio antiguo que sobre excavaciones, momias y pirámides, que es lo que yo esperaba.” Desengañado, cambió la arqueología por la medicina y después comenzó su doctorado en inmunología molecular.
Sin embargo, su atracción por Egipto seguía. En los años 80 estaba comenzando la secuenciación del ADN. Seguramente, pensaba Svante Pääbo, a alguien ya se le habrá ocurrido analizar el ADN de las momias egipcias. “Sabía, por mis estudios de Egiptología que había miles de momias en museos y que se descubrían cientos de momias cada año en Egipto.

iStockphoto
Pero en la literatura científica no había nada sobre extracción de ADN de momias, así que comencé el trabajo yo sólo.” Como sabía que su director de tesis no se lo consentiría, buscó ayuda en su profesor de egiptología. El trabajo de laboratorio lo realizó por las noches y en fines de semana. El resultado fue el sueño de cualquier joven científico: un artículo en Nature (Pääbo, 1985).

encontró a ‘Ötzi’ en los Alpes
de Ötztal
Imagen cortesía del Museo de
Arqueología de Tirol del Sur
Al terminar su doctorado y con la baza de su artículo en Nature, Svante Pääbo se trasladó a la Universidad de California en Berkeley, EE.UU. Allí comenzó a darse cuenta de lo difícil que resultaba evitar la contaminación cuando se trabaja con muestras de ADN humano. Si el ADN que estaba secuenciando era el de la momia y procedía realmente del antiguo Egipto o si pertenecía a los antropólogos que habían trabajado antes con esa momia. Se centró en la extracción de ADN de otros especímenes antiguos como el lobo marsupial (tigre de Tasmania) ya extinguido o el moa, una especie de avestruz incapaz de volar.
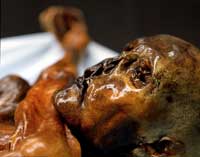
encontrado en buen estado
de conservación en los hielos
de los Alpes de Ötztal, en
1991
Imagen cortesía del Museo de
Arqueología de Tirol del Sur
En 1990, con 35 años, se trasladó a la Universidad de Múnich, Alemania, a ocupar una plaza de profesor y continuó trabajando con el ADN de organismos antiguos, incluyendo a los mamuts. La llegada de la PCR (reacción en cadena de la polimerasa) permitió a los investigadores replicar rápidamente muestras de ADN e hizo más fácil detectar la contaminación por ADN usando muestras de control. Esto animó al profesor Pääbo a retomar la investigación sobre el ADN humano en antiguos. En esta ocasión, formaba parte de un equipo que buscaba ADN de uno de los ‘antiguos egipcios’ contemporáneo de los europeos, ‘Ötzi’, el cuerpo de 5000 años de antigüedad en buen estado de conservación en los hielos de los Alpes de Ötztal, en la frontera austro-italiana en 1991.
Animado por el éxito en la investigación que demostró que el ADN mitocondrial de Ötzi (ver diagrama) era muy parecido al de las poblaciones del centro y norte de Europa (Handt et al., 1994), Svante Pääbo retrocedió más en el tiempo –unos 38000 años. Quería usar el análisis de ADN para investigar los orígenes humanos.
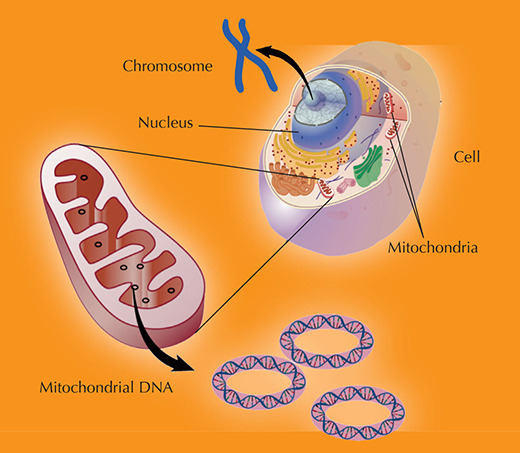
El ADN nuclear es herencia de los padres, cada progenitor aporta un cromosoma de cada par, así la descendencia obtiene la mitad de sus cromosomas del padre y la otra mitad de la madre.
Sin embargo la mitocondria, y por tanto su ADN, pasa de la madre a su descendencia
Imagen cortesía de Darryl Leja, NHGRI / NIH
“Una de las grandes ideas de los últimos 20 años es que los humanos modernos salieron de África recientemente”, explica el profesor Pääbo. Aunque en África vive mucha menos gente que fuera de África, “encontramos que la mayoría de las variaciones están presentes en África y que cualquiera de fuera de África es a su vez una variante de esa variación. En los últimos 100.000 años un grupo de africanos abandonó África y colonizó el resto del mundo. A mí me gusta decir que desde el punto de vista de la genética molecular, todos somos africanos: o vivimos en África o salimos de allí recientemente.”

encontraron los Neandertales
y los modernos humanos?
Imagen cortesía del Museo
Neanderthal, Alemania
Pero los humanos modernos, Homo sapiens, no eran los únicos humanos. Nuestros parientes, los neandertales, H. neanderthalensis, estaban moviéndose por Europa y Asia Occidental (el cercano oriente y oriente medio) desde, aproximadamente, el año 300.000 al año 30.000. Desde entonces, el descubrimiento en 1865 de algunos huesos humanos con apariencia muy antigua en el valle Neander, cerca de Düsseldorf, Alemania, ha surgido la controversia sobre el destino de los Neandertales.Cuando los humanos actuales migraron desde África a Europa, ¿exterminaron a los Neandertales?, ¿les vencieron o por el contrario se cruzaron entre ellos? Svante Pääbo y sus colegas decidieron averiguarlo comparando ADN extraído de los huesos de un ejemplar de Neanderthal de 38.000 años de antigüedad con los de varias poblaciones de modernos humanos.
Estos resultados eran fascinantes: los humanos modernos no africanos tenemos fragmentos de ADN similares a los de los Neandertales pero que no se encuentran en los africanos. De hecho, los datos científicos sugieren que entre el 1% y el 4% del genoma de los no africanos procede de los Neandertales (Noonan et al., 2006).
“La explicación más sencilla es que cuando los primeros humanos salieron de África, fueron hacia Oriente Medio y desde allí siguieron colonizando el resto del mundo. En Oriente Medio los humanos modernos se mezclaron con los Neandertales y sus descendientes incorporaron esas secuencias de ADN y las llevaron a Australia o Papúa-Nueva Guinea o a América”, explica el profesor Pääbo. Sólo la población africana se quedó sin ADN neanderthal (ver el diagrama inferior).
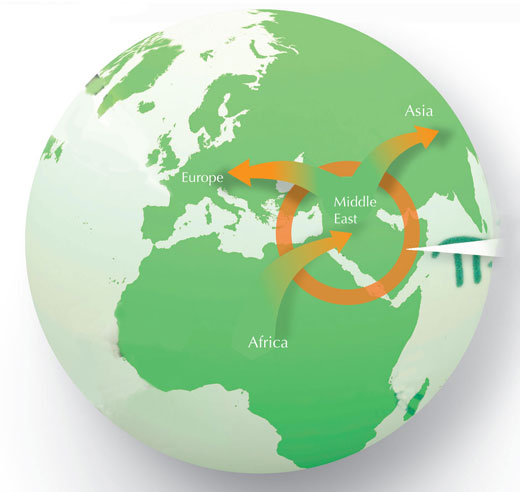
Imagen cortesía de Nicola Graf

Arqueología de Tirol del Sur /
Kennis / Ochsenknecht
“Lo que más me fascina es que analizando secuencias de ADN podemos contestar preguntas que no se pueden investigar mediante el estudio de los esqueletos de los primeros humanos o de las herramientas de piedra que fabricaron”
Desde 1997 y debido a su temprana fascinación por los orígenes del hombre, el profesor Pääbo ha sido director del Instituto Max Planck de Antropología Evolucionaria en Leipzig, Alemania.
Allí colaboran investigadores del campo de las ciencias y de las humanidades para investigar la historia de la humanidad comparando genes, cultura, habilidades cognitivas, lenguajes y sistemas sociales del pasado y presente de las poblaciones humanas y de los primates próximos a los humanos.
Le pregunto sobre su trabajo como investigador. “Trabajo en el laboratorio pero colaboramos con los que trabajan en excavaciones o los conservadores de los museos que reciben los restos de huesos. Cada vez más, podemos mirar no sólo a una parte del ADN sino a genomas completos; es crucial para poder analizar los datos con ordenadores.” Svante Pääbo, un investigador a pie de laboratorio encuentra esto un poco frustrante. “No conozco las bases de la bioinformática, por eso estoy en manos de esos expertos. Ahora, cuando formamos a los estudiantes nos aseguramos que no descuiden ninguna faceta: los investigadores jóvenes que trabajan fundamentalmente con ordenadores tienen que saber qué están analizando y viceversa.”
@EIROforum: investigación en la ESRF

Uno de los usos más interesantes de las fuentes de rayos-X de alta luminosidad producidos en la European Synchrotron Radiation Facility (ESRF)w2 es el estudio no invasivo de los fósiles. Los dientes fosilizados contienen información esencial y pueden usarse para determinar la edad exacta de un joven, cuándo murió, analizando el crecimiento diario de las líneas de esmalte dental.
Uno de los descubrimientos más importantes es que los neandertales alcanzaban la madurez más rápidamente que los modernos humanos, H. sapiens, más incluso que los que dejaron África hace unos 90.000-100.000 años. El patrón encontrado en los neandertales, H. neanderthalensis, parece ser un intermedio entre los encontrados en los primeros miembros del género, (por ejemplo, H. erectus) y los modernos humanos, (H. sapiens). Esto sugiere que el desarrollo lento y una larga infancia son característicos del H. sapiens y que el género Homo cambió desde una forma primitiva de ‘vivir rápido y procrear joven’ a ‘crecer despacio y aprender de tus padres’, estrategia que ha ayudado a que los humanos alcancen la situación actual en el planeta.
Para más información, ver las notas de prensaw3 o los artículos científicos originales, Macchiarelli et al. (2006) y Smith et al. (2010).
ESRF es uno de los miembros de EIROforumw4, editor de Science in School.
Actualmente el professor Pääbo trabaja en un pariente humano aún más lejano que los neandertales. A primera vista, su grupo no tenía muy poco con lo que continuar: apenas un fragmento de un pequeño hueso de un dedo encontrado en el sur de Siberia. Los científicos sabían que pertenecía a algún tipo de homínido –o humano- pero inicialmente sabían muy poco más. ¿Qué aspecto tenía?, ¿estaba muy extendido?, ¿podía ser un tipo de homínido encontrado a lo largo de Asia mientras que los neandertales se restringían a las regiones más occidentales?

Antón / PLoS Biology
Comenzaron secuenciando el genoma mitocondrial y descubrieron que era muy diferente tanto de los humanos modernos como de los neandertales. Comparando secuencias de ADN de los tres grupos, fueron capaces de estimar hace cuánto tiempo comenzaron a evolucionar por caminos separados. (Para saber cómo trabajar esto en el aula, ver Kozlowski, 2010.)
Mientras que el último antecesor común del neandertal y los humanos modernos vivió hace medio millón de años, el misterioso homínido se separó del Neandertal y del antecesor humano hace un millón de años. Aquel pequeño fragmento de hueso pertenecía a un pariente de los humanos bastante alejado.
Esta es la primera vez que un homínido nuevo se ha descrito basándose únicamente en el ADN pero, en opinión de Svante Pääbo, estos análisis irán siendo cada vez más habituales. “Este pequeño fragmento de hueso apenas si tiene información sobre la apariencia de su dueño, pero si se conserva suficientemente bien, nuestro siguiente paso será reconstruir el genoma completo a partir de este fragmento. Creo que en el futuro describiremos los organismos que descubramos más por su ADN que por su apariencia”.
Este artículo está basado en una entrevista con Svante Pääbo en el European Molecular Biology Laboratory en junio de 2010.
References
- Handt O et al. (1994) Molecular genetic analyses of the Tyrolean ice man. Science 264(5166): 1775-1778. doi: 10.1126/science.8209259
- Kozlowski C (2010) Bioinformatics with pen and paper: building a phylogenetic tree. Science in School 17: 28-33. www.scienceinschool.org/2010/issue17/bioinformatics
- Macchiarelli R et al. (2006) How Neanderthal molar teeth grew. Nature 444: 748-751. doi:10.1038/nature05314
-
Descarga gratis el artículo en este enlace, o suscríbete hoy a Nature: : www.nature.com/subscribe
-
- Noonan JP et al. (2006) Sequencing and analysis of Neanderthal genomic DNA. Science 314(5802): 1113-1118. doi: 10.1126/science.1131412
- Pääbo S (1985) Molecular cloning of ancient Egyptian mummy DNA. Nature 314(6012): 644-645. doi: 10.1038/314644a0
-
Descarga gratis el artículo en este enlace, o subscríbete hoy a Nature: www.nature.com/subscribe
-
- Rau M (2010) La ciencia es una aventura humana colectiva: entrevista con Pierre Léna. Science in School 14: 10-15. www.scienceinschool.org/2010/issue14/pierrelena/spanish
- Smith TM et al. (2010) Dental evidence for ontogenetic differences between modern humans and Neanderthals. Proceedings of the National Academy of Sciences of the United States of America 107(49): 20923-20928. doi: 10.1073/pnas.1010906107
-
Este artículo está disponible en la red.
-
Web References
- w1 – Puedes realizar una visita virtual a las nuevas instalaciones del European Molecular Biology Laboratory (EMBL), el laboratorio europeo de referencia en investigación básica en biología molecular que está en Heidelberg, Alemania. Ver: www.embl.de/events/atc/tour
-
EMBL es miembro de EIROforumw4, editor de Science in School. Para más información, ver www.embl.org
-
- w2 – El ESRF es una gran instalación científica internacional en Grenoble, Francia. Produce rayos X de alta luminosidad que son utilizados cada año por miles de científicos de todo el mundo. Para saber más: www.esrf.eu
- w3 – Los comunicados de prensa sobre la investigación en el ESRF de los Neandertales, están disponibles en la web del ESRFw2 o en estos enlaces http://tinyurl.com/neanderteeth y http://tinyurl.com/neanderkids
- w4 – Para más infomación sobre el EIROforum, ver: www.eiroforum.org
Resources
- Para saber más sobre los trabajos de Svante Pääbo sobre la evolución de los primates, ver el diálogo de ficción entre Demócrito, filósofo de la antigua Grecia y su discípulo: www.embl.de/aboutus/science_society/writing_prize/2002_jekely_brochure.pdf
- Para saber más sobre cómo nuestro ADN puede darnos información acerca de la evolución humana, ver los siguientes artículos de un estudiante de doctorado de Svante Pääbo:
-
Bryk J (2010) Selección natural a nivel molecular. Science in School 14: 58-62. www.scienceinschool.org/2010/issue14/evolution/spanish
-
Bryk J (2010) Evolución humana: comprobando las bases moleculares. Science in School 17: 11-16. www.scienceinschool.org/2010/issue17/evolution/spanish
-
- Una reseña sobre un relato de ficción sobre los Neandertales, ver:
-
Madden D (2008) Review of Dance of the Tiger. Science in School 8: 68. www.scienceinschool.org/2008/issue8/dance
-
- Para saber cómo las investigaciones con rayos X del ESRF puede arrojar luz sobre la evolución y las migraciones de nuestros antepasados lejanos, ver:
-
Tafforeau P (2007) Synchrotron light illuminates the orang-utan’s obscure origins. Science in School 5: 24-27. www.scienceinschool.org/2007/issue5/orangutan
-
- Más información sobre ‘Ötzi’, en la web del South Tyrol Museum of Archaeology, donde se conserva el hombre de hielo: www.iceman.it
Review
La ciencia de vanguardia no trata solo del futuro – la tecnología moderna también profundiza en la comprensión del pasado. Este artículo sobre cómo el estudio del genoma contribuye a la investigación arqueológica puede ser usado en clases de biología para interesar a los alumnos sobre genética y sobre evolución. ‘Ötzi’ también puede ser el punto de partida de una investigación interdisciplinar, que relacione la historia y la biología.
Algunas preguntas para evaluar la comprensión del artículo o de ampliación.
- Usar tus conocimientos sobre la estructura del ADN para comentar la arquitectura inspirada en la estructura del ADN
- Explicar la tecnología PCR.
- Explicar la evidencia de la afirmación “desde un punto de vista de genética molecular, todos somos africanos”.
- Explicar cómo secuenciaron los científicos el genoma mitocondrial. ¿Por qué se centraron el genoma mitocondrial en lugar de en del núcleo?
El artículo también puede ser usado para fomentar discusiones, por ejemplo sobre:
- La importancia de replica los experimentos y los controles (por qué la PCR es tan importante)
- El desarrollo del conocimiento científico (de una persona o de la humanidad – ver también Rau, 2010)
- La interdisciplinariedad de la ciencia
- Ética (a pesar de diferencias como el color de la piel, ‘todos somos africanos’).
Betina da Silva Lopes, Portugal





