Archäologe des Genoms: Svante Pääbo Understand article
Übersetzt von Hildegard Kienzle-Pfeilsticker. Der Evolutionsgenetiker Svante Pääbo erzählt Eleanor Hayes wie er das Genom ergründet, um die menschliche Evolution zu verstehen.

Training Centre am
Europäischen
Molekularbiologische
Laboratorium in Heidelberg,
Deutschland. Kann man die
Ähnlichkeiten zur
DNA-Doppelhelix erkennen?
Zum Vergrößern auf das Bild
klicken
Mit freundlicher Genehmigung
des EMBL Photolab
Ich hatte mir Archäologen immer bis zu den Knien im Matsch vorgestellt, wo sie Knochen, Gefäßscherben oder alten Schmuck ausgraben. Als ich aber Svante Pääbo traf, einen „Archäologen des Genoms“, war er überhaupt nicht verschlammt, noch waren wir draußen an der freien Luft.
Stattdessen trafen wir uns im glänzend neuen Advanced Training Centre am Europäischen Molekularbiologischen Laboratorium in Heidelberg, Deutschland. Das einem DNA-Molekül nachempfundene Gebäude hat zwei helikale Bürokorridore und verbindende Glasbrücken, die die Wasserstoffbrücken darstellen sollenw1.
Warum gibt es die Verbindung zwischen DNA und Archäologie? „In gewisser Weise machen meine Kollegen und ich das, was Archäologen machen“, erklärt Professor Pääbo. „Ausgrabungen, nicht in einer alten Höhle, sondern in unserem Genom. Wir untersuchen unsere DNA-Sequenzen nach Spuren unserer Geschichte, beispielsweise um herauszufinden, woher wir kamen und wie wir mit andern Menschentypen Kontakt hatten.“
Ich frage, wie er in dieses Forschungsfeld hineingeraten ist? Er erklärt, dass ihn eine frühe Faszination für Ägypten, die durch seine Mutter bei dortigen Urlauben befördert wurde, veranlasst hat, Ägyptologie in Uppsala, Schweden, zu studieren. Dort wurden allerdings seine romantischen Träume zerschlagen: „Wir lernten etwas über alte ägyptische Wortformen, statt Mumien und Pyramiden auszugraben, wie ich es mir vorgestellt hatte.“ Enttäuscht gab er auf und entschied sich, stattdessen Medizin zu studieren und dann begann er mit einer PhD-Arbeit in molekularer Immunologie.
Ägypten ließ mich jedoch nicht los. In den 1980er Jahren wurden die ersten DNA-Sequenzanalysen gemacht. Bestimmt hat jemand versucht, DNA aus alten ägyptischen Mumien zu extrahieren und zu analysieren, dachte Svante Pääbo. „Aus meinem Ägyptologiestudium wusste ich, dass Tausende von Mumien in Museen vorhanden sind und jedes Jahr weitere Hunderte in Ägypten entdeckt werden.

von sculpies / iStockphoto
Aber scheinbar gab es in der Literatur nichts über DNA-Extraktion aus ihnen, daher begann ich selbst daran zu arbeiten.” Da er wusste, dass sein Doktorvater nicht zustimmen würde, nahm er Hilfe von seinem früheren Ägyptologieprofessor an und arbeitete nachts und am Wochenende. Das Ergebnis war der Traum jedes jungen Wissenschaftlers: eine Veröffentlichung in Nature (Pääbo, 1985).

von „Ötzi“ in den Ötztaler
Alpen
Mit freundlicher Genehmigung
des Südtiroler
Archäologiemuseums
Nach seinem PhD und unterstützt durch seine Nature-Veröffentlichung ging Svante Pääbo an die Universität von California in Berkeley, USA. Dort begann er zu realisieren, wie schwierig es ist, bei der Arbeit mit alten menschlichen Proben Kontaminationen zu vermeiden – war die DNA, die er aus den Mumien extrahiert hatte, wirklich alte ägyptische DNA oder hatte er die DNA früherer Anthropologen sequenziert? Daher konzentrierte er sich darauf, DNA aus anderen, sehr alten Proben zu extrahieren, inklusive dem ausgestorbenen Beutelwolf und des Moa – einem riesigen, flügellosen Vogel.
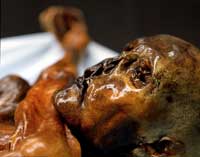
Körper, der im Eis
konserviert in den Ötztaler
Alpen 1991 gefunden wurde
Mit freundlicher Genehmigung
des Südtiroler
Archäologiemuseums.
Als er 1990 an die Universität München, Deutschland, ging, um mit nur 35 Jahren eine Professur anzunehmen, arbeitete er weiter über DNA aus ausgestorbenen Organismen, inklusive Mammuts. Die Erfindung der Polymerase-Kettenreaktion (PCR) ermöglichte es Wissenschaftlern, DNA-Proben schnell zu vervielfachen und erleichterte es, auf DNA-Kontaminationen zu prüfen, indem man viele identische Experimente und Kontrollen durchführte. Das ermutigte Professor Pääbo ein weiteres Mal, mit alter menschlicher DNA zu arbeiten. Dieses Mal war er Mitglied im Team, das die DNA eines der europäischen Zeitgenossen der alten Ägypter untersuchte: „Ötzi“, der 5000 Jahre alte Körper, der konserviert im Eis der Ötztaler Alpen an der österreichisch-italienischen Grenze 1991 gefunden wurde.
Inspiriert vom Erfolg der Forschung, die ergab, dass Ötzi’s mitochondriale DNA (siehe Diagramm unten) der der modernen zentral- und nordeuropäischen Populationen ähnlich war (Handt et al., 1994), begab sich Svante Pääbo weiter zurück in der Zeit – etwa 38 000 Jahre in die Vergangenheit. Er wollte mittels DNA-Analyse die menschliche Herkunft untersuchen.
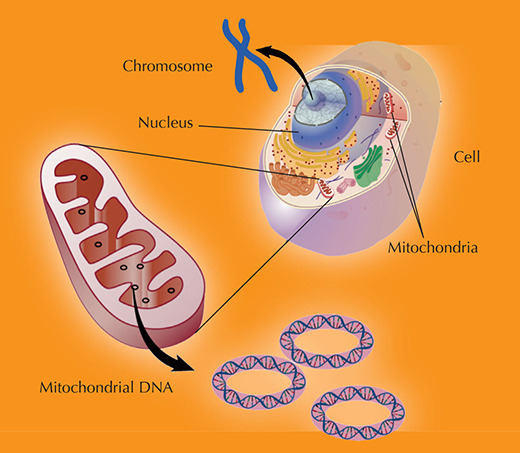
Nukleäre DNA wird von beiden Eltern geerbt: jeder Elternteil trägt ein Chromosom zu jedem Paar bei, so dass die Nachkommen die eine Hälfte der Chromosomen vom Vater und die andere Hälfte von der Mutter bekommen.
Im Gegensatz dazu werden Mitochondrien und damit mitochondriale DNA von der Mutter an die Nachkommen weitergegeben
Mit freundlicher Genehmigung von Darryl Leja, NHGRI / NIH
“Eine der größten Erkenntnisse der letzten 20 Jahre auf diesem Gebiet ist, dass moderne Menschen vor nicht allzu langer Zeit aus Afrika kamen“, erklärt Professor Pääbo. Obwohl in Afrika viel weniger Leute leben als außerhalb, „bei der Untersuchung menschlicher DNA-Sequenzen finden wir, dass die meisten Varianten in Afrika existieren und jeder außerhalb von Afrika zu einer dieser Varianten gehört. Es stellt sich heraus, dass während der letzten 100 000 Jahre eine Gruppe Afrikaner Afrika verlassen und den Rest der Welt besiedelt hat. Ich möchte daher formulieren, dass wir vom molekulargenetischen Standpunkt aus alle Afrikaner sind; wir leben entweder in Afrika oder sind erst vor Kurzem ins Exil gegangen.“

Zusammentreffen moderner
Menschen mit Neandertalern?
Mit freundlicher Genehmigung
des Neanderthal Museums,
Deutschland
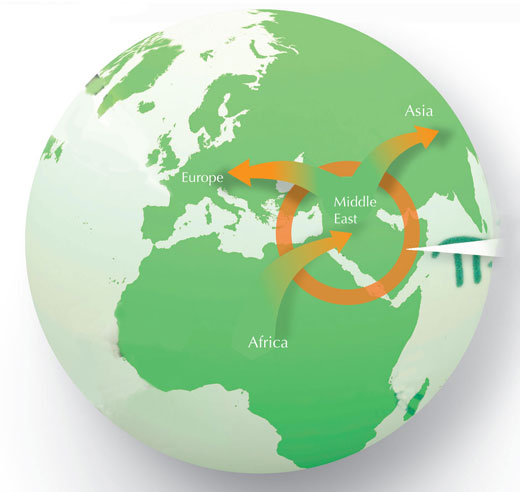
Aber moderne Menschen, Homo sapiens, waren zu dieser Zeit nicht die einzigen Menschen auf der Welt. In Europa und Westasien (im Nahen und Mittleren Osten) waren vor 300 000 bis 30 000 Jahren unsere Verwandten unterwegs: die Neandertaler, H. neanderthalensis. Seit der Entdeckung einiger seltsam aussehender, menschlicher Knochen im Jahr 1856 im Neandertal nahe Düsseldorf, Deutschland, gab es viele Kontroversen über das Schicksal der Neandertaler. Töteten moderne Menschen den Neandertaler, als sie von Afrika nach Europa wanderten? Haben sie ihn aus dem Feld geschlagen? Oder haben sie sich mit fortgepflanzt? Wenn wir Europäer ein Bild von einem Neandertaler sehen: blicken wir auf einen unserer frühen Vorfahren? Svante Pääbo und seine Kollegen wollten es herausfinden, indem sie extrahierte DNA aus den Knochen eines 38 000 Jahre alten Neandertalers mit DNA-Proben mehrerer Populationen moderner Menschen verglichen.
Die Ergebnisse waren faszinierend: Moderne Menschen außerhalb Afrikas tragen einige Sequenzen, die den Sequenzen der Neandertaler ähneln, aber nicht in Afrikanern zu finden sind. Die Daten der Wissenschaftler legen tatsächlich nahe, dass zwischen 1 und 4% des Genoms der Nicht-Afrikaner von Neandertalern abstammt (Noonan et al., 2006).
“Die einfachste Erklärung ist, dass moderne Menschen, als sie das erste Mal Afrika verließen, durch den Mittleren Osten kamen und dann weiterwanderten und den Rest der Welt besiedelten. Im Mittleren Osten pflanzten sich moderne Menschen mit Neandertalern fort und ihre Abkömmlinge trugen die DNA-Sequenzen der Neandertaler mit sich – nach Australien oder Papua-Neuguinea oder nach Amerika“, erklärt Professor Pääbeo. Nur die afrikanischen Populationen blieben unberührt (siehe das unten stehende Diagramm).
Mit freundlicher Genehmigung von Nicola Graf

des Südtiroler
Archäologiemuseums / Kennis
/ Ochsenknecht
“Was mich so fasziniert, ist, dass wir mit DNA-Sequenzanalyse Fragen beantworten können, die wir durch die Begutachtung der Skelette früher Menschen oder deren hinterlassener Steinwerkzeuge nicht angehen können.“
Getreu seiner frühen Faszination für menschliche Herkunft war Professor Pääbo seit 1997 Direktor des Max-Planck-Instituts für evolutionäre Anthropologie in Leipzig, Deutschland.
Dort untersuchen Forscher aus den Natur- und Geisteswissenschaften die Geschichte der Menschheit durch Vergleiche von Genen, Kulturen, kognitiven Fähigkeiten, Sprachen und Sozialsystemen vergangener und gegenwärtiger menschlicher Populationen, ebenso wie von den Menschen nahe verwandten Primaten.
Wie, frage ich ihn, macht er seine Arbeit? „Ich arbeite im Labor, aber wir kooperieren mit den Leuten, die die Knochen ausgraben oder mit den Museumskuratoren, die die Knochen erhalten. Und es wird zunehmend wichtiger, die Daten mit Computern zu analysieren, weil wir die Möglichkeit haben, nicht nur kleine Bereiche der DNA, sondern ganze Genome zu untersuchen.“ Svante Pääbo, ein praktischer Laborforscher, findet das manchmal frustrierend. „Ich kenne das A und O der Bioinformatik nicht wirklich, daher bin ich abhängig von Leuten, die das verstehen. Wenn wir heute unsere Studenten ausbilden, stellen wir sicher, dass sie beide Seiten kennen lernen: junge Wissenschaftler, die in erster Linie mit Computern arbeiten, müssen einige Zeit im Labor verbringen, um zu verstehen, was passiert, und umgekehrt.“
@EIROforum: ESRF Forschung

Eine der spannendsten Anwendungen der sehr intensiven Röntgenstrahlen, die am European Synchrotron Radiation Facility (ESRF)w2 generiert werden, ist die nicht-invasive Untersuchung von Fossilien. Besonders fossile Zähne enthalten wichtige Informationen, weil sie dazu benutzt werden können, das exakte Alter eines Jugendlichen bei seinem Tod zu bestimmen: durch zählen der täglichen Wachstumslinien im Zahnschmelz.
Eine bemerkenswerte Erkenntnis neuerer Studien ist, dass Neandertaler wesentlich schneller als moderne Menschen, H. sapiens, inklusive einige der frühesten Gruppen, die Afrika vor 90-100 000 Jahren verlassen haben, erwachsen wurden. Das in Neandertalern, H. neanderthalensis, gefundene Muster scheint zwischen dem in frühen Vertretern der Gattung (z.B. H. erectus) und dem moderner Menschen (H. sapiens) zu liegen. Das lässt vermuten, dass langsame Entwicklung und lange Kindheit Charakteristika von H. sapiens sind, und dass die Gattung Homo von einem einfachen “lebe schnell und pflanze dich jung fort“-Zustand zu einer „wachse langsam und lerne von deinen Eltern“-Strategie übergegangen ist, welche modernen Menschen geholfen hat, ihre derzeitige Stellung auf unserem Planeten einzunehmen.
Mehr dazu finden Sie in den Pressemitteilungenw3 oder in der Originalliteratur: Macchiarelli et al. (2006) und Smith et al. (2010).
ESRF ist eines der Mitglieder von EIROforumw4, dem Herausgeber von Science in School.

von Mauricio Antón / PLoS
Biology
Zur Zeit arbeitet Professor Pääbo an einem noch weiter verwandten menschlichen Verwandten als die Neandertaler. Und auf den ersten Blick hatten er und seine Kollegen wenig Material zur Verfügung: nur die Spitze eines kleinen Fingerknochens, der in Südsibirien gefunden worden war. Die Wissenschaftler wussten, dass dieser Knochen zu einer Art Hominide gehört – oder einer menschlichen Form – wussten aber anfangs sehr wenig über ihn. Wie sah er aus? Könnte er ein Hominiden-Typ gewesen sein, den man in ganz Asien antreffen konnte, während die Neandertaler auf mehr westliche Regionen beschränkt waren?
Die Wissenschaftler begannen mit der Sequenzierung des mitochondrialen Genoms und fanden, dass es sich sehr von dem sowohl moderner Menschen als auch von Neandertalern unterschied. Durch Vergleich der DNA-Sequenzen der drei Gruppen konnten sie abschätzen, vor wie langer Zeit sie sich getrennt weiterentwickelt haben. (Wie man das im Unterricht nachvollziehen kann siehe Kozlowski, 2010.)
Während der letzte gemeinsame Vorfahr von Neandertalern und modernem Menschen bis vor einer halben Million Jahre lebte, spaltete sich der mysteriöse Hominide vom Neandertaler-Vorfahren vor etwa einer Million Jahre ab. Das kleine Knochenfragment gehörte tatsächlich zu einem sehr entfernten, menschlichen Verwandten.
Dies ist das erste Mal, dass ein neuer Hominide ausschließlich durch seine DNA-Sequenz beschrieben wurde, aber, das glaubt Svante Pääbo, solche Analysen werden immer populärer werden. „Dieses kleine Knochenfragment trägt kaum irgendwelche Informationen über das Aussehen des Individuums, aber wenn es gut genug erhalten ist, wird unser nächster Schritt sein, das gesamte Genom daraus zu rekonstruieren. Ich denke, dass wir künftig neu entdeckte Organismen eher mit ihrer DNA, denn mit ihrem Aussehen beschreiben werden.“
Dieser Artikel basiert auf einem Interview mit Svante Pääbo am Europäischen Molekularbiologischen Laboratorium im Juni 2010.
References
- Handt O et al. (1994) Molecular genetic analyses of the Tyrolean ice man. Science 264(5166): 1775-1778. doi: 10.1126/science.8209259
- Kozlowski C (2010) Bioinformatik mit Stift und Papier: wie man einen phylogenetischen Stammbaum aufstellt. Science in School 17. www.scienceinschool.org/2010/issue17/bioinformatics/german
- Macchiarelli R et al. (2006) How Neanderthal molar teeth grew. Nature 444: 748-751. doi:10.1038/nature05314
-
Laden Sie den Artikel hier kostenlos herunter, oder abonnieren Sie noch heute die Zeitschrift Nature: www.nature.com/subscribe
-
- Noonan JP et al. (2006) Sequencing and analysis of Neanderthal genomic DNA. Science 314(5802): 1113-1118. doi: 10.1126/science.1131412
- Pääbo S (1985) Molecular cloning of ancient Egyptian mummy DNA. Nature 314(6012): 644-645. doi: 10.1038/314644a0
-
Laden Sie den Artikel hier kostenlos herunter, oder abonnieren Sie noch heute die Zeitschrift Nature: www.nature.com/subscribe
-
- Rau M (2010) Science is a collective human adventure: interview with Pierre Léna. Science in School 14: 10-15. www.scienceinschool.org/2010/issue14/pierrelena
- Smith TM et al. (2010) Dental evidence for ontogenetic differences between modern humans and Neanderthals. Proceedings of the National Academy of Sciences of the United States of America 107(49): 20923-20928. doi: 10.1073/pnas.1010906107
-
Dieser Open-Access-Artikel ist online frei zugänglich.
-
Web References
- w1 – Für eine virtuelle Tour durch das neue Gebäude am Europäischen Molekularbiologischen Laboratorium (EMBL), das europäische Flaggschiff-Labor für Grundlagenforschung in der Molekularbiologie in Heidelberg, Deutschland, siehe: www.embl.de/events/atc/tour
-
EMBL ist Mitglied im EIROforum w4, dem Herausgeber von Science in School. Mehr erfahren Sie unter: www.embl.org
-
- w2 – Ein internationales Forschungszentrum in Grenoble, Frankreich, das ESRF, produziert Röntgenstrahlen hoher Brillianz, die Tausenden von Wissenschaftlern auf der ganzen Welt jedes Jahr nutzen. Mehr dazu erfahren Sie unter: www.esrf.eu
- w3 – Die Pressemeldungen des ESRF über Neandertalerforschung sind auf der ESRF-Webseite verfügbar w2oder über die direkten Links: http://tinyurl.com/neanderteeth und http://tinyurl.com/neanderkids
- w4 – Zu mehr Informationen über das EIROforum siehe: www.eiroforum.org
Resources
- Um mehr über Svante Pääbos Arbeit über die Primatenevolution zu erfahren, siehe den unterhaltsamen, fiktiven Dialog zwischendem alten griechischen Philosophen Demokrit und seinem Studenten: www.embl.de/aboutus/science_society/writing_prize/2002_jekely_brochure.pdf
- Mehr dazu, was uns DNA über die menschliche Evolution verraten kann, siehe die folgenden Artikel von einem der letzten PhD-Studenten von Svante Pääbo:
-
Bryk J (2010) Natürliche Selektion auf molekularer Ebene. Science in School 14. www.scienceinschool.org/2010/issue14/evolution/german
-
Bryk J (2010) Die Evolution des Menschen auf molekularer Ebene Science in School 17. www.scienceinschool.org/2010/issue17/evolution/german
-
- Für einen Review über eine fiktive Beschreibung der Neandertaler siehe:
-
Madden D (2008) Review of Dance of the Tiger. Science in School 8: 68. www.scienceinschool.org/2008/issue8/dance
-
- Wie Untersuchungen mit Röntgenstrahlen am ESRF Licht auf die Evolution und Wanderung entfernterer menschlicher Vorfahren werfen können, siehe:
-
Tafforeau P (2007) Synchrotron light illuminates the orang-utan’s obscure origins. Science in School 5: 24-27. www.scienceinschool.org/2007/issue5/orangutan
-
- Zu weiteren Informationen über „Ötzi” siehe die Webseite des Südtiroler Archäologiemuseums, in dem der Eismann konserviert wird:www.iceman.it
Review
Moderne Wissenschaft dreht sich nicht nur um die Zukunft – moderne Technologie kann auch das Verständnis der Vergangenheit vertiefen. Dieser Artikel beschreibt, wie das Studium des Genoms zur archäologischen Forschung im Biologieunterricht beiträgt, um das Interesse von Schülern für Genetik und Evolution zu wecken. Er könnte auch der Ausgangspunkt interdisziplinärer Forschung sein, die Biologie und Geschichte verbindet, vielleicht am Beispiel „Ötzi“.
Geeignete Verständnis- und Ergänzungsfragen beeinhalten:
- Kommentieren Sie anhand Ihres Wissens über die DNA-Struktur deren Einfluss auf die Gestaltbildung.
- Erklären Sie die PCR- Technologie.
- Erklären Sie die Evidenz für die Behauptung “aus molekulargenetischer Sichtweise sind wir alle Afrikaner”.
- Erklären Sie, wie Wissenschaftler das Mitochondriengenom sequenziert haben. Warum haben sie sich auf das mitochondriale statt auf das Genom des Zellkerns konzentriert?
Der Artikel könnte auch als Ausgangspunkt von Diskussionen genutzt werden, zum Beispiel über:
- die Bedeutung wiederholter Experimente und von Kontrollen (warum PCR so wichtig war),
- die Entwicklung wissenschaftlichen Wissens (für ein Individuum oder die Menschen im Allgemeinen – siehe auch Rau, 2010)
- die Interdisziplinarität von Wissenschaft,
- Ethik (trotz Unterschieden in der Hautfarbe „sind wir alle Afrikaner“).
Betina da Silva Lopes, Portugal





