Svante Pääbo, où l archéologie du génome Understand article
Traduit par Claire Debrat. Le chercheur en génétique évolutionniste Svante Pääbo explique à Eleanor Hayes comment il fouille le génome pour pour comprendre l'évolution humaine.

au Laboratoire Européen de
Biologie Moléculaire à
Heidelberg, en Allemagne,
tout juste construit.
Pouvez-vous voir la
ressemblance avec la double
hélice de l’ADN ? Cliquer sur
l’image pour l’agrandir
Image reproduite avec
l’aimable autorisation de l’EMBL
Photolab
J’ai toujours imaginé trouver des archéologues en train de déterrer des os, des fragments de poterie ou des bijoux anciens, avec de la boue jusqu’aux genoux. Pourtant, quand j’ai rencontré Svante Pääbo, un « archéologue du génome », il n’était absolument pas couvert de boue et nous n’étions pas en plein air.
À la place, nous nous sommes rencontrés – comme il se doit – dans le flambant neuf Advanced Training Centre du Laboratoire Européen de Biologie Moléculaire, à Heidelberg, en Allemagne. Ce bâtiment, conçu pour ressembler à une hélice d’ADN, est constitué de deux couloirs de bureaux en hélice, connectés par des ponts de verre qui représentent les liaisons hydrogènew1.
Pourquoi ce lien entre ADN et archéologie? « D’un certaine façon, mes collègues et moi-même faisons la même chose que des archéologues », explique le professeur Pääbo. « Nous ne fouillons pas d’anciennes grottes, mais le génome. Nous scrutons les traces de notre histoire dans nos séquences d’ADN, par exemple pour comprendre d’où nous venons et comment nous avons interagi avec d’autres types d’humains ».
Je lui demande comment il est arrivé dans ce domaine de recherche. Il explique que, après des vacances passées avec sa mère en Égypte, une fascination pour le pays l’a pris et poussé à étudier l’Égyptologie à l’Université de Uppsala, en Suède. Une fois dedans, cependant, ses rêves de romantique furent écrasés : « Nous étudiions les anciennes formes d’écriture égyptienne, au lieu de déterrer des momies et fouiller des pyramides en Égypte, comme je l’avais imaginé ». Déçu, il décide de se tourner à la place vers la médecine, puis se lance dans un Doctorat en Immunologie Moléculaire.
L’attrait pour l’Égypte est pourtant toujours bien là. Dans les années 80, l’analyse des séquences ADN commence tout juste. Il y a forcément quelqu’un, pense Svante Pääbo, qui aura tenté d’extraire et analyser l’ADN des momies égyptiennes. « Je savais, grâce à mes études en Égyptologie, que des centaines de momies se trouvaient dans des musées et que d’autres centaines étaient découvertes chaque années en Égypte.

l’aimable autorisation de
sculpies / iStockphoto
Comme il n’y avait apparemment rien dans les publications parlant d’en extraire l’ADN, j’ai commencé à travailler dessus moi-même ». Certain que son directeur de thèse ne le suivra pas, il demande de l’aide à son ancien professeur d’Égyptologie et fait le travail de laboratoire la nuit et le weekend. Le résultat: le rêve de tout jeune scientifique – un article dans Nature (Pääbo, 1985).

découvert ‘Ötzi’ Dans les
Alpes de l’Ötztal
Image reproduite avec
l’aimable autorisation du South
Tyrol Museum of Archaeology
Au sortir de son Doctorat, et avec le sésame de son article dans Nature, Svante Pääbo déménage à l’Université de Californie à Berkely, USA. C’est là qu’il se rend compte de la difficulté d’éviter les contaminations lorsque l’on travaille sur des échantillons humains anciens – l’ADN extrait est-il celui de la momie ou celui des anthropologistes ? Il se concentre alors sur l’extraction d’ADN d’autres spécimens anciens, parmi lesquels celui des espèces éteintes du loup de Tasmanie et du moa – un oiseau géant qui ne volait pas.
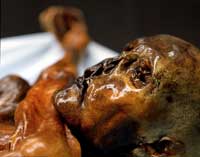
retrouvé conservé dans la
glace dans les Alpes de
l’Ötztal, en 1991
Image reproduite avec
l’aimable autorisation du South
Tyrol Museum of Archaeology
Il continue de travailler sur l’ADN d’anciens organismes, tel que le mammouth, après son déménagement à l’Université de Munich, en Allemagne, où, à seulement 35 ans, il a obtenu une chaire de professeur. L’avènement de la PCR (Amplification en Chaîne par Polymérase) permet aux scientifiques d’amplifier rapidement des échantillons d’ADN et de vérifier plus simplement la présence de contamination, en faisant plusieurs expériences simultanées et en utilisant des contrôles. Ce qui encourage le professeur Pääbo a reprendre ses travaux sur d’anciens échantillons humains. Ce coup-ci, il fait partie d’une équipe qui étudie l’ADN d’un contemporain européen des anciens Égyptiens : ‘Ötzi’, un corps vieux de 5000 ans retrouvé conservé dans la glace, en 1991, dans les Alpes de l’Ötztal, à la frontière Austro-Italienne.
Encouragé par la réussite du projet, qui démontre que l’ADN mitochondrial (voir illustration ci-dessous) d’Ötzi est très proche de celui des populations modernes d’Europe centrale et du nord (Handt et al., 1994), Svante Pääbo remonte encore plus loin dans le temps – à environ 38.000 ans. Il veut interroger l’ADN sur les origines de l’Homme.
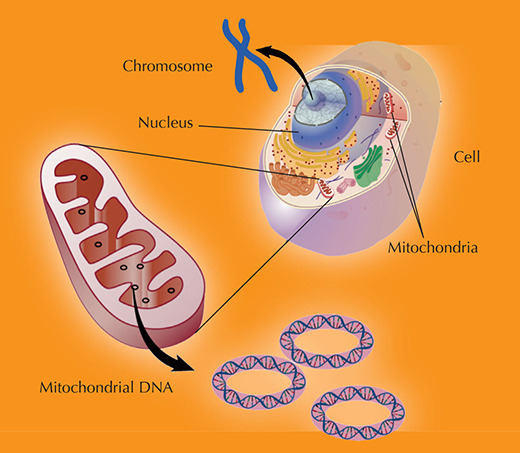
L’ADN nucléaire est hérité des deux parents : chaque parent fourni un chromosome de chaque paire, de sorte que le génome nucléaire dont la descendance hérite provient pour moitié de la mère et pour moitié du père.
Les mitochondries, et donc l’ADN mitochondrial, en revanche, sont transmis par la mère à la descendance
Image reproduite avec l’aimable autorisation de Darryl Leja, NHGRI / NIH
« Dans ce domaine, un des grands pas de ces dernières 20 années est que l’Homme moderne est venu d’Afrique assez récemment », explique le professeur Pääbo. Bien qu’il y ait bien moins de personnes vivant en Afrique que hors d’Afrique, « si l’on étudie les variations de séquences ADN chez les humains, on s’aperçoit que la plupart des variations existent en Afrique, tandis que tout le monde hors d’Afrique ne présente qu’une partie de ces variations. Il semble s’avérer que, au cours des 100.000 dernières années, un groupe d’Africains a quitté l’Afrique et colonisé le reste du monde. C’est pourquoi j’aime dire que, du point de vue génomique, nous sommes tous Africains ; soit nous vivons en Afrique, soit nous avons émigré depuis peu ».

Hommes modernes sont
entrés en contact avec les
Hommes de Néandertal?
Image reproduite avec
l’aimable autorisation du
Neanderthal Museum,
Allemagne
L’Homme moderne, Homo sapiens, n’était cependant pas le seul Homme existant à l’époque. Entre il y a 300.000 et 30.000 ans environ, nos « cousins », les Hommes de Néandertal – H. neanderthalensis – parcouraient l’Europe et l’Asie occidentale. Depuis la découverte, en 1856, d’ossements humains à l’aspect inhabituel dans la Vallée de Neander, près de Düsseldorf, en Allemagne, la question du sort des Néandertaliens est l’objet d’une vive controverse. Les Hommes modernes ont-ils tué les Hommes de Néandertal lorsqu’ils ont émigré d’Afrique en Europe ? Les ont-ils supplantés ? Ou se sont-ils mélangés et reproduits avec eux ? Lorsqu’un Européen regarde une image d’un Néandertalien, est-il en train de regarder un lointain ancêtre ? Svante Pääbo et ses collègues ont décidé d’en avoir le cœur net en comparant les extraits d’ADN venant d’ossements d’un spécimen Néandertalien de 38.000 ans et celui de différentes populations d’Hommes modernes.
Les résultats sont fascinants: les Hommes modernes non-Africains présentent certaines séquences ADN similaires à des séquences que l’on trouve chez Néandertal, mais pas les Africains. De fait, les données des scientifiques suggèrent que entre 1 et 4% du génome des non-Africains est d’origine néandertalienne (Noonan et al., 2006).
« L’explication la plus simple est que, quittant l’Afrique, les premiers humains modernes soient arrivées dans le Moyen-Orient et, de là, aient poursuivi vers le reste du monde. Au Moyen-Orient, les Hommes modernes se seraient reproduits avec des Néandertaliens, ce qui fait que leurs descendants portaient en eux, et auraient emporté avec eux, des séquences ADN de Néandertal – jusqu’en Australie, Papouasie-Nouvelle-Guinée ou dans les Amériques », explique le professeur Pääbo. Seules les populations africaines n’auraient pas été affectées (voir le schéma ci-dessous).
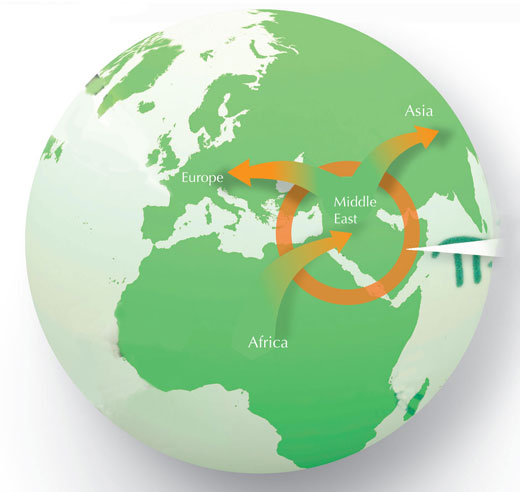
Image reproduite avec l’aimable autorisation de Nicola Graf

l’aimable autorisation du South
Tyrol Museum of Archaeology /
Kennis / Ochsenknecht
« Ce qui me fascine, c’est que l’analyse des séquences ADN nous permet de répondre à des questions auxquelles l’observation des squelettes des premiers Hommes, ou des outils en pierre qu’ils ont laissé derrière eux, ne permet pas de répondre.”
Fidèle à sa fascination pour les origines de l’Homme, le professeur Pääbo est depuis 1997 directeur de l’Institut Max Planck pour l’Anthropologie Évolutionniste à Leipzig, en Allemagne.
Là, des chercheurs en sciences et sciences humaines scrutent l’Histoire de l’humanité en comparant les gènes, les cultures, les capacités cognitives, les langues et les systèmes sociaux de populations humaines passées et présentes, ainsi que celles des primates les plus proches des humains.
Je lui demande comment s’effectuent ses recherches. « Mon travail est au laboratoire, mais nous collaborons avec les gens qui extraient les ossements ou avec les conservateurs de musées qui reçoivent les ossements. De plus en plus, du fait que l’on peut étudier non seulement des petits segments d’ADN, mais aussi des génomes entiers, il est indispensable d’analyser les données par ordinateurs. Svante Pääbo, qui aime contrôler sa recherche en laboratoire, trouve parfois cela un peu frustrant. « Je ne connait pas vraiment les rouages de la bioinformatique, donc je suis entre les mains des gens qui les connaissent. Aujourd’hui, lorsque nous formons nos étudiants, nous nous assurons qu’ils apprennent les deux aspects: les jeunes scientifiques qui travaillent principalement sur ordinateur ont besoin de passer quelques temps en laboratoire pour comprendre ce qui s’y passe, et vice versa ».
@EIROforum: la recherche à l’ESRF

Les rayons X intenses produis à l’ESRFw2 offrent une possibilité excitante pour l’étude des fossiles : ils sont non-invasifs (i.e ils n’abîment pas l’échantillon). Les dents fossilisées, en particulier, renferment des informations essentielles car elles peuvent servir à déterminer l’âge exact auquel un jeune est mort – en comptant les lignes de croissance journalières de l’émail.
Des études récentes ont permit une découverte notable : un Néandertalien atteignait l’âge adulte plus rapidement qu’un H.sapiens (y compris les premiers à avoir quitté le continent Africain, il y a 90-100.000 ans), et cette différence est significative. Les caractéristiques de l’Homme de Néandertal, H.neanderthalensis, semblent correspondre à un intermédiaire entre celles des membres primitif du genre (ex: H.erectus) et l’Homme moderne (H.sapiens). Ceci suggère qu’un développement lent et une enfance longue sont des caractéristiques d’H.sapiens, et que le genre Homo a glissé d’une condition primitive où il fallait « vivre vite et se reproduire tôt » vers une stratégie de « grandir lentement et apprendre des parents », laquelle a permis aux Hommes modernes de conquérir leur position actuelle sur la planète.
Pour en savoir plus, voir les dépêchesw3 ou consulter les articles originaux : Macchiarelli et al. (2006) et Smith et al. (2010).
L’ESRF est membre du EIROforumw4, l’éditeur de Science in School.

l’aimable autorisation de
Mauricio Antón / PLoS Biology
Actuellement, le professeur Pääbo travaille sur un cousin humain encore plus éloigné que Néandertal. À première vue, lui et ses collègues n’ont pas grand chose pour progresser: à peine le bout d’un os d’auriculaire, découvert dans le sud de la Sibérie. Les scientifiques savaient que cet os appartenait à une sorte d’hominidé – cad une forme d’humain – mais, au départ, ils en savaient très peu. À quoi ressemblait-il ? Était-il très répandu ? Était-ce un type d’hominidé réparti d’un bout à l’autre de l’Asie tandis que Néandertal était confiné aux régions les plus occidentales ?
Les scientifiques ont commencé à séquencer le génome mitochondrial, et ont découvert qu’il était très différent à la fois de celui de l’Homme moderne et de l’Homme de Néandertal. En comparant les séquences ADN des trois groupes, ils ont réussi à estimer le moment dans le temps où ils ont commencé à évoluer séparément. (Pour apprendre à faire cela en classe, voir Kozlowski, 2010.)
Alors que le dernier ancêtre commun à l’Homme moderne et à l’Homme de Néandertal a vécu il y a 500.000 ans, le mystérieux hominidé a divergé de cet ancêtre il y a environ un million d’années. Ce petit fragment osseux appartenait à un très lointain membre de la famille!
C’est la première fois qu’un nouvel hominidé est décrit uniquement par sa séquence ADN, mais Svante Pääbo est convaincu que de telles analyses vont devenir de plus en plus populaires. « Ce petit fragment d’os fourni très peu d’informations quant à l’aspect de l’individu mais, s’il est suffisamment conservé, la prochaine étape sera de reconstituer le génome entier à partir de cet échantillon. À l’avenir, je pense que l’on décrira les organismes récemment découverts pas leur ADN plutôt que par leur aspect ».
Une entrevue avec Svante Pääbo au Laboratoire Européen de Biologie Moléculaire, en juin 2010, a servi de base à cet article.
References
- Handt O et al. (1994) Molecular genetic analyses of the Tyrolean ice man. Science 264(5166): 1775-1778. doi: 10.1126/science.8209259
- Kozlowski C (2010) Bioinformatics with pen and paper: building a phylogenetic tree. Science in School 17: 28-33. www.scienceinschool.org/2010/issue17/bioinformatics
- Macchiarelli R et al. (2006) How Neanderthal molar teeth grew. Nature 444: 748-751. doi:10.1038/nature05314
-
Télécharger gratuitement l’article ici, ou s’abonner maintenant à Nature: www.nature.com/subscribe
-
- Noonan JP et al. (2006) Sequencing and analysis of Neanderthal genomic DNA. Science 314(5802): 1113-1118. doi: 10.1126/science.1131412
- Pääbo S (1985) Molecular cloning of ancient Egyptian mummy DNA. Nature 314(6012): 644-645. doi: 10.1038/314644a0
-
Télécharger gratuitement l’article ici, ou s’abonner maintenant à Nature: www.nature.com/subscribe
-
- Rau M (2010) La science est une aventure humaine collective : entrevue avec Pierre Léna. Science in School 14: 10-15. www.scienceinschool.org/2010/issue14/pierrelena/french
- Smith TM et al. (2010) Dental evidence for ontogenetic differences between modern humans and Neanderthals. Proceedings of the National Academy of Sciences of the United States of America 107(49): 20923-20928. doi: 10.1073/pnas.1010906107
-
Cet article en libre accès est disponible gratuitement sur internet.
-
Web References
- w1 – Pour effectuer une visite virtuelle du nouveau bâtiment à l’Institut Européen de Biologie Moléculaire (EMBL), le fleuron européen pour la recherche fondamentale en biologie moléculaire à Heidelberg, Allemagne, voir: www.embl.de/events/atc/tour
-
L’EMBL est membre du EIROforumw4 l’éditeur de Science in School. Pour en savoir plus, consultez www.embl.org
-
- w2 – L’ESRF, un centre de recherche international à Grenoble, en France, produit des rayons X extrêmement brillants, que des milliers de scientifiques du monde entier utilisent chaque année. Pour en savoir plus, voir: www.esrf.eu
- w3 – Les dépêches au sujet des recherches à l’ESRF sur Néandertal sont disponibles sur le site de l’ESRFw2 ou via les liens directs http://tinyurl.com/neanderteeth et http://tinyurl.com/neanderkids
- w4 – Informations supplémentaires à propos de EIROforum, voir: www.eiroforum.org
Resources
- Pour en savoir plus sur les travaux de Svante Pääbo sur l’évolution des primates, voir le dialogue fictif amusant entre le philosophe grec Démocrite et son étudiant: www.embl.de/aboutus/science_society/writing_prize/2002_jekely_brochure.pdf
- Pour en savoir plus sur ce que notre ADN peut nous apprendre sur notre évolution, voir les articles suivants d’un doctorant récent de Svante Pääbo:
-
Bryk J (2010) La sélection naturelle vue au niveau moléculaire. Science in School 14: 58-62. www.scienceinschool.org/2010/issue14/evolution/french
-
Bryk J (2010) Evolution humaine: tester l’hypothèse de la base moléculaire. Science in School 17: 11-16. www.scienceinschool.org/2010/issue17/evolution/french
-
- La critique d’un récit fictif sur l’Homme de Néandertal:
-
Madden D (2008) Review of Dance of the Tiger. Science in School 8: 68. www.scienceinschool.org/2008/issue8/dance
-
- Pour découvrir en quoi les recherches aux rayons X de l’ESRF peuvent nous éclairer sur l’évolution et la migration de nos lointains ancêtres, voir:
-
Tafforeau P (2007) La lumière synchrotron éclaire les origines obscures des orangs-outangs. Science in School 5: 24-27. www.scienceinschool.org/2007/issue5/orangutan/french
-
- Pour plus d’informations sur ‘Ötzi’, voir le site du Musée Archéologique du Tyrol du Sud, où le corps glacé est conservé: www.iceman.it
Review
La pointe de la science ne concerne pas seulement le futur – les technologies modernes peuvent aussi améliorer notre compréhension du passé. Cet article, qui montre l’apport des études du génome au domaine de l’archéologie, peut être utilisé en classe de biologie pour intéresser les étudiants à la génétique et à l’évolution. On peut aussi s’en servir comme point de départ pour faire des ponts entre biologie et histoire, peut-être en partant de « Ötzi ».
Exemples de questions de compréhension possibles, ou pour aller plus loin::
- En utilisant vos connaissances sur la structure de l’ADN, commenter l’architecture du bâtiment ayant la forme d’une molécule d’ADN.
- Expliquer la technologie de la PCR.
- Expliquer les preuves qui soutiennent l’affirmation « du point de vue de la génomique moléculaire, nous sommes tous Africains ».
- Expliquer comment les scientifiques ont séquencé l’ADN mitochondrial. Pourquoi ont-ils porté leur attention sur le génome mitochondrial plutôt que nucléaire?
Cet article peut aussi servir de point de départ pour des discussions sur des sujets tels que:
- L’importance de répéter les expérimentations et les contrôles (ou pourquoi la PCR est si importante)
- Le développement des connaissances scientifiques (pour un individu, ou plus généralement pour les humains – voir aussi Rau, 2010)
- Le fait que la science soit interdisciplinaire
- Éthique (malgré des différences, telles que la couleur de la peau, “nous sommes tous Africains”).
Betina da Silva Lopes, Portugal





