Uso de bases de datos biológicas para enseñar evolución y bioquímica Teach article
Traducido por Javier Aseguinolaza Iriondo. Se pueden utilizar herramientas online para comparar las secuencias de proteínas y entender lo diferente que ha sido la evolución de organismos.
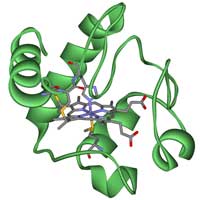
Citocromo c
Imagen cortesía de Klaus
Hoffmeier/Wikimedia commons
En el pasado, los científicos realizaron análisis de evolución comparando las características físicas de las especies – conocidas como sus fenotipos – que se encontraron en registros de fósiles. Sin embargo, todo esto cambió desde el descubrimiento del reloj molecular.
El concepto de reloj molecular surgió de la observación de que cuanto más tiempo pasa desde que dos especies divergen de un ancestro común, tanto más diferentes serán sus DNA o secuencias de sus proteínas (para revisar, Bromham & Penny, 2003).
Comparando genes homólogos o secuencias de proteínas – en otras palabras, aquellas de dos organismos con un ancestro común – puedes medir cuanto tiempo ha pasado desde que los organismos divergieran. Esto se puede visualizar en un árbol filogenético.
Para examinar cómo de similares son dos genes, hay que tener sus secuencias y alinearlas correctamente (Kozlowski, 2010). Era realmente difícil obtener esas secuencias, pero ya no.
Probablemente sus estudiantes le digan que todo está en internet – esta vez, tienen razón. Hay muchos ejemplos de bases de datos biológicas accesibles que contengan datos de investigaciones reales en internet, pero para esta actividad vamos a utilizar dos recursos en particular.
El Centro Nacional de Información Biotecnológica (NCBI)w1 de Bethesda, MD, EEUU, ofrece acceso a información biomédica y genómica, mientras que el Instituto Europeo de Bioinformática (EBI)w2, localizado en Hinxton, GB, ofrece datos accesibles gratuitamente de experimentos de ciencia de la vida y realiza investigación básica en biología computacional. La base de datos del NCBI te ofrecerá la secuencia de cualquier gen o proteína que ya haya sido secuenciada, y después usted puede utilizar herramientas del EBI para alinear las secuencias y analizarlas.
Actividad
Cuando se investigan relaciones de evolución entre organismos diferentes, es importante elegir con cuidado qué gen o molécula se usará. Hay algunos genes homólogos bien conocidos que se pueden utilizar, tales como los utilizados para las proteínas hemoglobina o citocromo c, usaremos éste último en esta actividad. El citocromo c es una hemo-proteína pequeña que es componente central de la cadena de transporte de electrones en la mitocondria. Todos los organismos aeróbicos han evolucionado de un ancestro común que utilizó primero citocromo c, por lo que es una buena elección para nuestras intencionesw3.
Esta actividad se realiza en tres secciones diferentes:
- encontrar la secuencia de aminoácido del citocromo c en diferentes organismos,
- alinearlos, y
- hacer un árbol filogenético.
Finalmente, se incluyen algunas cuesyiones para guiar la investigación de relaciones de evolución.

para aumentar
Encontrar secuencias de proteínas
- Ir a la página webw1 de NCBI.
- En la zona de búsqueda, en la parte superior de la página, seleccionar «proteína» en el menú desplegable.
- Escribe el nombre de una especie, ej: Homo sapiens, y citocromo c.
- Hacer clic en el botón de búsqueda.
- Una nueva página dará la lista de resultados. La mayor parte de ellos tienen la misma secuencia, a partir de fuentes diferentes, pero otros pueden ser secuencias parciales o pertenecer a una especie o proteína diferente. Elija con cuidado la proteína correcta de interés y haga clic en el enlace inferior marcado como ‘FASTA’.
- De la nueva página que se cargue, copie la cadena de letras mayúsculas que representa la secuencia de aminoácidos. Pegue las letras en un documento Word, recuerde etiquetar la secuencia con el nombre del organismo del que se obtuvo.
- Haga lo mismo con tantos organismos como desee, dependiendo de lo que quiera investigar con sus estudiantes. Puede incluir varios primates para ver cómo evolucionaron los humanos u organismos de los cinco reinos tradicionales para ver cómo evolucionó la vida en general. En esta actividad se utilizarán 3 animales, 2 plantas, 2 algas, un hongo y un protozoo.
Alineando secuencias
- Vaya al sitio webw2 del Instituto Europeo de Bioinformatica (EBI) y haga clic en «Servicios». Después seleccione «proteínas».
- Haga clic en Clustal Omega. Copie el texto de su focumento Word y peguelo en el cuadro de texto etiquetado como «PASO 1» (STEP 1).
- En el PASO 2 (STEP 2), elija un formato para la alineación de salida, tal como el ‘Clustal w/ numbers’ que mostrará la longitud de cada secuencia. Finalmente, haga clic en «entregar» para completar el PASO 3 (STEP 3).
- Las alineaciones para las múltiples secuencias aparecerán en una nueva ventana. Lo primero que puede hacer es hacer clic para mostrar los colores. Esta opción dará el mismo color a cada aminoácido, lo que los hace más fácilmente indentificables.
- Para analizar la alineación, tenga en mente los siguientes símbolos: un asterisco (*) significa que las secuencias son idénticas en esa posición; dos puntosa (:) indica sustituciones conservadas (mismo color de grupo); y un punto y seguido and (.) se refiere a una sustitución semi- conservada (formas similares). Los colores agrupan los aminoácidos por características. Los rojos son pequeños, hidrofóbicos, aromáticos; los azules son ácidos; los magenta son básicos; los verdes son hidroxilos, aminas, amidas, básicos; y los grises son el resto.
- Si hace clic en la opción «Resultado Resumen», puede ver el pocentaje de identidad conservado entre los diferentes organismos tras la alineación. En esta matriz, puede encontrar el porcentaje de identidad de dos organismos para la secuencia de la proteína citocromo c. Además, si tiene Java™ instalado en su ordenador, puede usar Jalview, un programa gratuito para editar múltiples alineaciones de secuencias, visualización y análisis. Con Jalview, podrá ver la secuencia consensuada del citocromo c y el nivel de conservación de los diferentes aminoácidos.
El software Clustal Omega tiene muchas opciones diferentes que involucran más sofisticado conocimiento del que necesitamos para nuestras intenciones. Si quiere saber más sobre el uso de Clustal Omega, vea el artículo de Sievers et al. (2011).
para aumentar
Hacer un árbol filogenético
- En los resultados de Clustal Omega, haga clic en «Arbol filogenético» en la parte inferior (necesitará tener instalado Java™).
- Puede obtener un árbol filogenético o cladograma. En un cladograma, las longitudes de las ramas del árbol son arbitrarias, mientras que en un árbol filogenético, las longitudes de las ramas indican cuánto ha evolucionado la proteína en el tiempo.
Para una discusión posterior
para aumentar
- Las moléculas homólogas son un ejemplo de evolución divergente. Como puede explicar la evolución divergente utilizando el citocromo c?
- Las alineaciones se pueden hacer utilizando secuencias de nucleótidos (genes) o aminoácidos (proteínas). Por qué cree que es más útil usar proteínas wue ADN para analizar relaciones de evolución?
- En arboles filogenéticos, un «clado» queda formado por todos los organismos que tienen un ancestro común. Dé un ejemplo de su cladograma.
- Qué organismos han sufrido un evento de especiación más recientemente, según análisis filogenéticos del citocromo c? Cuál es el número de eventos de especiación?
- Por qué cree que algunos aminoácidos han cambiado debido a mutación y otros no? Cree que los aminoácidos conservados no han cambiado porque sus codonantes no han sufrido ninguna mutación en absoluto?
- Muestre alguno de los aminoácidos conservados en su alineación. Investigue su función en Internet.
Glosario
Cladograma: Un diagrama ramificado que muestra la relación de evolución entre especies, con longitudes de rama arbitrarias.
Secuencia de consenso: Un conjunto conocido de secuencias conservadas, o el orden calculado de los aminoácidos más frecuentes encontrados en cada posición en una alineación de secuencias.
Aminoácido conservado: Una secuencia de aminoácidos en un polipéptido que es similar en múltiples organismos.
FASTA: un formato basado en texto para representar secuencias de nucleótidos o péptidos usando códigos de una sola letra. Una secuencia en formato FASTA comienza con una descripción en una sola línea, seguida por líneas de datos de secuencias. La línea de descripción se distingue de los datos de secuencia a través del símbolo mayor que (>).
Proteína homóloga: Aquellas proteínas compartidas por algunos organismos que derivan de un ancestro común.
Arbol filogenético: Un diagrama ramificado que muestra la relación de evolución entre especies, donde las longitudes de las ramas indican la diferencia entre las dos proteínas o genes.
Evento de Especiación: El momento en que especies ancestrales divergen hacia nuevas especies.
Agradecimientos
El autor querría agradecer a su colega María Isern su ayuda en la revisión de la gramática inglesa de este artículo.
References
- Bromhan L. Penny D. (2003) The modern molecular clock. Nature Reviews Genetics 5: 216-224
- Kozlowski C. (2010) La bioinformática con papel y lápiz: construcción de un árbol filogenético. Science in School 17: 28-33.
- Sievers F. et al. (2011) Fast, scalable generation of high-quality protein multiple sequence alignments using Clustal Omega. Molecular Systems Biology 7: 539
- Este articulo está disponible gratuitamente en la web de Molecular Systems Biology (Biología de Sistemas Moleculares).
Web References
- w1 – El Centro Nacional De EEUU para la Información Biotecnológica (US National Center for Biotechnology Information) ofrece acceso a información biomédica y genómica.
- w2 – El Instituto Europeo de Bioinformática (European Bioinformatics Institute) ofrece datos accesibles gratuitamente, de experimentos de ciencias de la vida, realiza investigación básica en biología computacional y ofrece un amplio programa de formación a usuarios, apoyando a los investigadores en el ámbito académico y en el industrial.
- w3 – John Kimball ha escrito un libro de biología disponible online denominado ‘Taxonomía: Clasificando la Vida’ que incluye un capítulo sobre ‘Árboles filogenéticos’.
Resources
- El sitio web ‘Understanding Evolution’ (entendiendo la evolución) del Museo de Paleontología de la Universidad de California ofrece muy Buena información sobre la construcción y lectura de árboles filogenéticos.
- Para aprender más sobre el uso del citocromo c en árboles fologenéticos.
- El sitio web Protein Data Bank (banco de datos de proteínas) en Europa pertenece al Instituto Europeo de Bioinformática y puede utilizarse para buscar y ver la estructura 3D del citocromo c.
- El Tree of Life website (sitio web del árbol de la vida) te permite explorar interactivamente y ver las series de televisión del Arbol de la Vida que la BBC transmitió con Sir David Attenborough.
Review
Los profesores de Biología podrían utilizar este artículo para enlazar temss de biología de la evolución, historia de la ciencia, bioquímica y genética. Para aprovechar este artículo, es importante que los estudiantes entiendan los fundamentos del ADN y la bioquímica de las proteínas.
La actividad descrita en el artículo es importante para motivar a los estudiantes a trabajar con autonomía en investigación real usando bases de datos científicas. Dentro del aula de laboratorio, los estudiantes podrían ser guiados trabajando en pequeños grupos, comparando secuencias de proteínas, tales como el citocromo c, o DNA, para comprender las diferencias entre los árboles filognéticos y el cladograma. La bioinformática es muy útil en la escuela secundaria para llevar a cabo “un aprendizaje integrado del contenido y del lenguaje, a diferentes niveles, con profesores de inglés, historia y física en un proyecto interdisciplinar. Este artículo, que enlaza estas diferentes asignaturas, también podría abrir discusiones sobre el progreso y las limitaciones de tal investigación.
Marina Minoli, Experta Didáctica del Centro Experto de la Universidad de Agora, Italia






