La evolución en acción: los patógenos Understand article
Los patógenos que amenazan la salud humana evolucionan constantemente para mantenerse en una posición de ventaja frente a nuestras defensas. Pero ahora podemos observar estos cambios a nivel genético, incluso mientras ocurren.
Una de las principales fuerzas impulsoras de la evolución es la selección natural, por la cual unos organismos individuales con una determinada estructura genética se reproducen más que otros en un medio dado. Eso da lugar a adaptaciones en ese organismo que le permiten sobrevivir, mientras otros organismos perecen.
Science Photo Library/De Angelis, Maurizio
Como estableció Darwin, este proceso de selección natural positiva (en el que los rasgos que fortalecen la aptitud de un organismo se convierten en dominantes en una población) contribuye a la diversidad que vemos en las especies vivas. Pero también constituye un factor en procesos que amenazan nuestra propia supervivencia, como la propagación de las infecciones víricas y la aparición de la resistencia a los antibióticos en las bacterias patógenas. Sin embargo, gracias a ingeniosos experimentos de laboratorio (véase, por ejemplo, Bryk, 2017), y a los avances de la tecnología de la secuenciación de genes, como los secuenciadores portátiles, ahora podemos observar este proceso de manera rápida y precisa, no solo en el laboratorio, sino en cualquier lugar del mundo. Esto ha resultado sumamente valioso para comprender la evolución de los microorganismos que causan enfermedades.
En este artículo comentamos dos extraordinarios estudios que examinan cómo evolucionan constantemente los patógenos y revelan los patrones del cambio genético detrás de este proceso, junto con los factores que lo limitan.
Bajo la lupa: la resistencia a los antibióticos
Un ejemplo clásico de evolución por selección natural positiva es la aparición de la resistencia a los antibióticos en las bacterias. El uso de antibióticos ejerce una presión selectiva sobre una población bacteriana, que muta y se reproduce muy rápidamente. En consecuencia, las variantes genéticas que protegen a las bacterias de la acción de los antibióticos permanecen en la población, y todas las otras variantes desaparecen.
Michael Baym, investigador de la universidad de Harvard (Estados Unidos) y autoproclamado luchador contra la resistencia a los antibióticos, ha documentado este proceso con una cámara de video. En 2015, Baym creó una forma de visualizar cómo se propagan las bacterias resistentes en un medio con antibiótico. Usó una enorme placa de Petri rectangular, de 60 cm de ancho por 120 cm de largo (una placa de Petri estándar mide 9 cm de diámetro), llena de agar (un medio mucilaginoso en el que las bacterias pueden crecer) teñido de negro. Inoculó el agar con concentraciones de antibiótico que iban aumentando en etapas a lo largo de la placa (Figura 1), desde cero unidades de antibiótico en los extremos hasta 1000 en las secciones del centro, concentración que debería ser suficiente para matar a cualquier bacteria. Instaló una luz y una cámara arriba de la placa para que las colonias bacterianas pudieran verse como puntos blancos contra el fondo negro del agar. Luego, sembró la bacteria Escherichia coli (E. coli) en los extremos de la placa, donde no había antibiótico y esperó que el proceso comience (Figura 2).

Laboratorio Kishony (Harvard Medical School) y Technion (Instituto de Tecnología de Israel)
¿Y qué ocurrió? La bacteria E. coli se podía desplazar en la capa superior del agar, así que cuando se le acabó el alimento en su propio vecindario, se mudó a otra región (Figura 3). Sin embargo, el antibiótico del área vecina impidió su crecimiento, por lo que solamente la bacteria con mutaciones que le permitía sobrevivir a esa exposición podía propagarse. Al comienzo algunos mutantes invadieron la primera región que contenía el antibiótico (de solo una unidad de concentración). Ellos se reprodujeron y la generación siguiente se extendió por toda esa región, hasta que ellos mismos se encontraron con una mayor concentración de antibiótico de la región subsiguiente. En los límites de las regiones, la bacteria hacía una pausa hasta que aparecían nuevas mutaciones con una mayor tolerancia al antibiótico.
Este proceso se repitió en los límites de cada región hasta que, luego de 11 días de crecimiento bacteriano, la bacteria E. coli cubrió toda la superficie de la enorme placa. Las bacterias en el centro de la placa habían creado una resistencia a concentraciones de antibiótico 1000 veces mayor a las que estaban cerca de los bordes (Figura 4). Se registró cada paso de este proceso en un video que ahora es muy conocido w1.

Laboratorio Kishony (Harvard Medical School) y Technion (Instituto de Tecnología de Israel)

Laboratorio Kishony (Harvard Medical School) y Technion (Instituto de Tecnología de Israel)

Laboratorio Kishony (Harvard Medical School) y Technion (Instituto de Tecnología de Israel)
Como los investigadores podían ver la propagación de las bacterias en cada etapa, también podían obtener una muestra de las bacterias en las zonas cruciales donde se originaban determinados mutantes. Al secuenciar estos genomas, lograron identificar los cambios a cada paso de la evolución de la resistencia (Figura 5).
Resultó ser que muchas de las mutaciones ocurrieron en múltiples ocasiones, lo que indicaría que algunos caminos a la resistencia son más comunes que otros. Además, algunas mutaciones ocurrieron en genes que aparentemente no tenían nada que ver con el antibiótico, lo que ilustró una cuestión importante del desarrollo de la resistencia: la necesidad de adaptarse al antibiótico constituye una carga para los procesos metabólicos básicos, por lo que en ausencia de antibióticos, las bacterias resistentes a menudo crecen en forma más lenta que las no resistentes. Necesitan las mutaciones adicionales para compensar la carga metabólica, y esto puede ser otro motivo de la pausa de crecimiento en los límites de las regiones donde hay una mayor concentración de antibiótico.
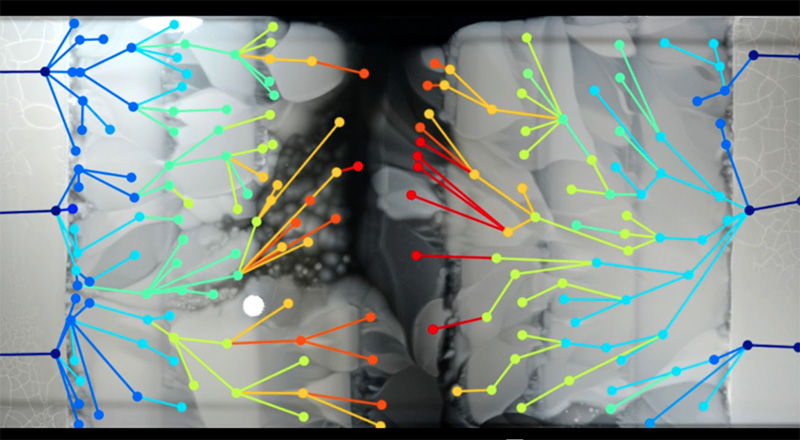
Laboratorio Kishony (Harvard Medical School) y Technion (Instituto de Tecnología de Israel)
Posteriormente, Baym y sus colegas hicieron otra observación crucial. Si hacían que la diferencia entre las concentraciones de antibiótico de las regiones vecinas fuera mayor, los mutantes no podrían sobrevivir en la región subsiguiente y el crecimiento bacteriano se detendría por completo en el primer límite entre regiones. Este fenómeno puede explicarse con el mismo principio: cuando la presión selectiva que los antibióticos ejercen sobre el medio es muy alta, es casi imposible que la bacteria crezca, porque los mecanismos que se necesitan para superar ese obstáculo requieren demasiadas mutaciones al mismo tiempo; entonces, las bacterias mueren.
La epidemia de ébola
Otro ejemplo de la evolución de los patógenos microbianos que se ha estudiado de manera intensiva es uno de los patógenos humanos más mortales: el virus del Ébola. La epidemia de ébola de 2013 a 2015 fue la de mayor duración y alcance hasta ahora, con 28 646 casos y 11 323 muertes registradas hacia junio de 2016. La duración de la epidemia, junto con la llegada de la tecnología de secuenciación de genes que podía usarse en el mismo sitio, permitió que los investigadores observen la evolución del virus mientras este infectaba a nuevos pacientes.
Los genetistas con base en Estados Unidos
Pardis Sabeti (Universidad de Harvard), Jeremy Luban (Facultad de Medicina, Universidad de Massachusetts) y Andrew Rambaut (Universidad de Edinburgo) lideraron grupos de científicos que estudiaban el ébola. Los científicos construyeron un «árbol genealógico» del virus mientras este se propagaba por África occidental a lo largo de dos años (Park, 2015).Cada rama del árbol representaba una nueva serie de mutaciones que le permitían al virus multiplicarse y crecer mejor que otras cepas. Al comparar los genomas víricos recogidos de pacientes en diferentes países y en diferentes momentos, el grupo determinó que la variante principal del virus se había originado en Guinea y que había logrado propagarse luego de adquirir solamente cinco mutaciones nuevas. Mientras se extendía de Guinea a Sierra Leona en mayo de 2014, apareció otra mutación y se volvió mucho más común en esa región (Diehl et al., 2016)w2. La nueva mutación se observó por primera vez en un solo paciente, pero fue tan exitosa que el 97 % de los más de 200 genomas del ébola analizados en todo el estudio fueron descendientes de ese mutante. Es la extraordinaria y exitosa historia de una cepa que había surgido hacía solo dos años (Figura 6).
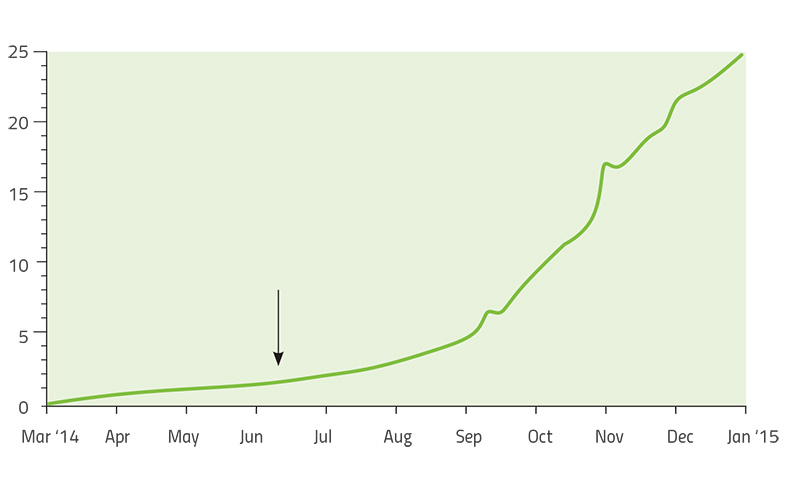
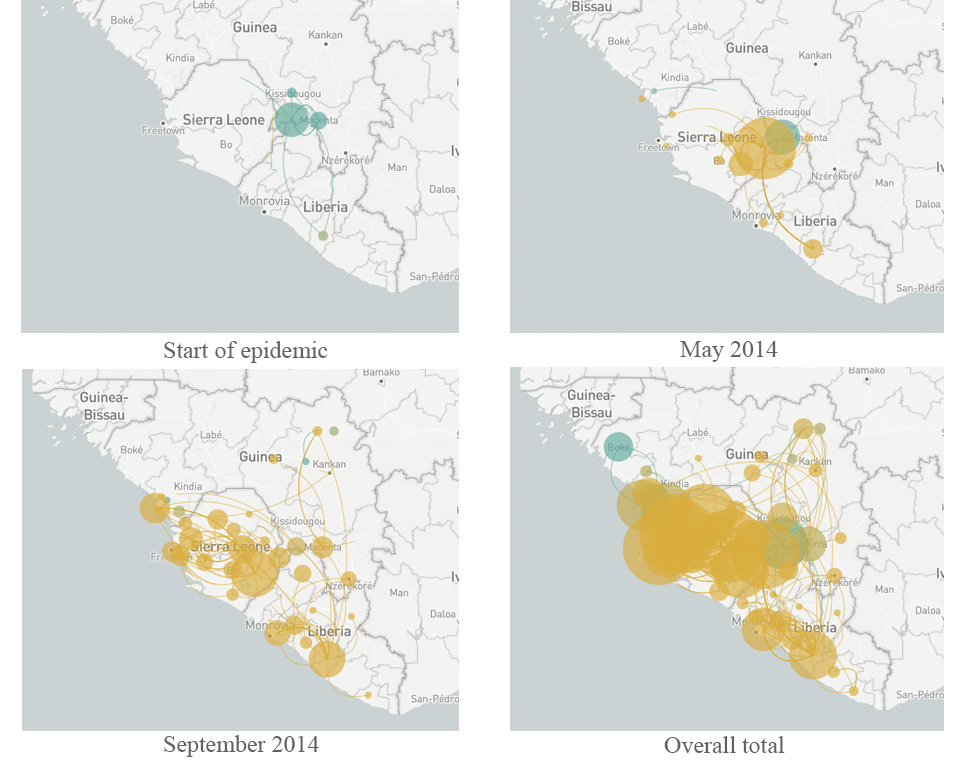
(Fuente: nextstrain.org)
Significativamente, el estudio permitió observar algunos genes del genoma del ébola que habían cambiado mucho más de lo esperado, incluidos los de las moléculas de proteínas que estaban ubicadas fuera del virus. Se piensa que estas proteínas víricas son un blanco para los anticuerpos humanos que luchan contra la infección; los rápidos cambios en estas proteínas ayudarían al virus a evitar que el anfitrión lo neutralice; por lo tanto, se trata de un modelo basado en la selección natural. En la investigación actual, estos genes han adquirido una importancia prometedora para contrarrestar la propagación del virus en una futura epidemia. Investigar los mecanismos de la evolución también ayuda a los científicos a desarrollar estrategias efectivas para combatir los patógenos en el futuro.
References
- Bryk J (2017) Evolution in action: the 67 000-generation experiment. Science in School 41: 24-29.
- Park D et al. (2015) Ebola virus epidemiology, transmission, and evolution during seven months in Sierra Leone. Cell 161 1516-1526. doi: 10.1016/j.cell.2015.06.007
- Diehl W E et al. (2016) Ebola virus glycoprotein with increased infectivity dominated the 2013–2016 epidemic. Cell 167 1088–1097. doi: 10.1016/j.cell.2016.10.014
Web References
- w1 – Puede ver el video de Michael Baym y sus colegas que muestra cómo se propaga la bacteria E. coli por las regiones con concentraciones cada vez mayores de antibiótico en la placa gigante.
- w2 – Puede ver una animación que muestra la propagación del virus del Ébola y cómo lo afectó la nueva cepa que surgió aproximadamente en junio de 2014.
Resources
- En el sitio web The Atlantic puede consultar un artículo accesible sobre el experimento de Baym y sus implicaciones.
- Puede leer un artículo científico en la revista Science sobre el experimento de Baym: Baym M. et al. (2016).
- Baym M et al. (2016) Spatiotemporal microbial evolution on antibiotic landscapes. Science 53: 1147-1151. doi: 10.1126/science.aag0822
- Puede ver la charla TED sobre la lucha contra el ébola de la investigadora Pardis Sabeti.
- Puede leer la entrada en un blog sobre la labor de observación del virus del Ébola, con enlaces a la cobertura en los medios y a la tecnología que se empleó.
- Puede encontrar una guía sobre el ébola en el sitio web Médecins Sans Frontière.
Review
Uno de los principales problemas al enseñar la evolución es la dimensión del tiempo. Debido a que la vida humana es corta en comparación con los períodos de tiempo en que ocurren los grandes procesos evolutivos, el concepto de cambio a lo largo del tiempo presenta una dificultad básica para comprender la evolución. Por ello, los ejemplos de procesos evolutivos que ocurren en un período de tiempo compatible con la percepción humana brindan oportunidades para superar esas dificultades.
El experimento que describe este artículo ofrece un buen panorama de cómo distintos elementos como el tiempo y las mutaciones afectan la evolución. Los docentes pueden usar este artículo para enseñar la evolución y para demostrar que esta puede ocurrir durante un período corto de tiempo. Además, el artículo sirve para explicar el papel central de las mutaciones en la evolución como causante de la diversidad sobre la que luego actúa la selección natural. Y como el experimento incluye una idea conocida (el uso de antibióticos), es fácil de entender.
Panagiotis K. Stasinakis, docente de biología del cuarto año de la escuela secundaria de Zografou (Grecia)





