Das Aufdecken unseres genetischen Bauplans Understand article
Übersetzt von Verena Müller. Was macht der Großteil unserer DNA? Hunderte Wissenschaftler untersuchen seit Jahren diese “Müll”-Sequenzen, die der Schlüssel zu schweren Krankheiten sein könnten - und noch viel mehr.

von fr73 / iStockphoto
Das Humangenomprojekt – die Sequenzierung des menschlichen Genoms – war eine der bedeutendsten Errungenschaften des letzten Jahrzehnts: der genetische Bauplan des Menschen, drei Milliarden Basen, wurde aufgedeckt, aber die Geschichte war noch nicht zu Ende. Um die Funktionsweise des Genoms zu verstehen, muss man zunächst entschlüsseln, wie die Sequenz von unseren Zellen interpretiert wird. Erst dann können wir dieses Wissen vielleicht in der biomedizinischen Forschung und dem Gesundheitswesen anwenden.

automatischen
Sequenziermaschine, wie sie
im Humangenomprojekt
verwendet wurde, um die
gesamte DNA-Sequenz des
Menschen zu bestimmen.
Jeder Peak steht für eine
bestimmte Base. Das
Humangenomprojekt hat drei
Milliarden Buchstaben
identifiziert, welche unser
Genom bilden. ENCODE
liefert uns nun Details, wie
das Genom funktioniert. Zum
Vergrößern auf das Bild
klicken.
Mit freundlicher Genehmigung
von Genome Research Limited
Eine der größten Überraschungen des menschlichen Genoms war, dass nur 2 % des Genoms auch Gene enthalten, die Baupläne für Proteine. Nachdem man andere Elemente des Genoms, wie nicht-kodierende RNAs, die die Aktivität von Genen und Introns (der Teil einer Gensequenz, welcher entfernt wird, bevor eine Boten-RNA übersetzt wird) kontrollieren, berücksichtigt hatte, war die Allgemeinheit der Ansicht, dass der Rest des Genoms keine biologische Funktion hat. Dies hatte zur Folge, dass dieser Rest häufig als “Müll”-DNA bezeichnet wurde.
Über die Sequenz hinaus
Als das Humangenom sequenziert war, war es an der Zeit herauszufinden, ob diese Sequenzen wirklich “Müll” waren. 2003 wurde das ENCODE-Konsortium gebildet, um die nichtkodierenden, aber funktionellen Teile des humanen Genoms zu charakterisieren. Das Konsortium wurde durch das “National Human Genome Research Institute” in den USA unterstützt und vom Europäischen Bioinformatik-Institut (EBI; siehe Kasten) in Großbritannien geleitet. In der ENCODE- Pilotphase von 2003 bis 2007 konnte ein globales Netzwerk an Wissenschaftlern experimentelle Methoden und Berechnungen zur Bestimmung der aktiven Bereiche in 1 % des Genoms testen, vergleichen und verbessern – im Prinzip haben sie einen Teil des genomischen “Mülls” durchsucht.
Mit freundlicher Genehmigung von Ian Dunham
Projekt hat 147 verschiedene
Zelltypen analysiert, um die
Unterschiede in der
Genregulation in
verschiedenen Gewebstypen
zu verstehen. Die Darstellung
zeigt 47 der 147
verschiedenen Zelltypen, die
in die Studie miteinbezogen
wurden. Es wurden viele
verschiedene Zelltypen
verwendet, da Zelltypen die
Informationen des Genoms
unterschiedlich nutzen,
obwohl sie alle dasselbe
Genom haben. Zum
Vergrößern auf das Bild
klicken.
Mit freundlicher Genehmigung
von Darryl Leja
Die ersten Ergebnisse wurden im Juni 2007 veröffentlicht (The ENCODE Project Consortium, 2007), und haben spannende Einblicke in die Tätigkeit des Genoms gewährt. Zum Beispiel haben die Daten aus Microarray- (siehe Koutsos et al., 2009) und Sequenzierungs-Experimenten gezeigt, dass der Großteil des Genoms transkribiert wird, auch manche Regionen, von denen man bis dahin gedacht hatte, dass sie inaktiv sind (Abbildung 2). Obwohl die biologische Funktion der meisten Transkripte noch nicht bekannt war, wurde gezeigt, dass manche wichtige Genexpressions-Regulatoren sind. Insgesamt hat diese Momentaufnahme des Genoms gezeigt, dass das Zusammenspiel der Gene mit Regionen, welche die Aktivität von Genen regulieren und anderen DNA-Sequenzen bei weitem komplexer ist, als bisher angenommen wurde. Die Daten haben schon da angedeutet, dass das Genom viele unterschiedliche, aktive Elemente beinhaltet und demnach auch weniger unbenutzte Sequenzen, als bisher gedacht wurde.
Die ENCODE-Wissenschaftler haben dann, als sie ihre Herangehensweise erfolgreich getestet hatten, damit angefangen, das ganze menschliche Genom zu untersuchen. Dies wurde durch Fortschritte in der Technologie der DNA-Sequenzierung und der Verfügbarkeit präziserer biologischer Assays begünstigt.
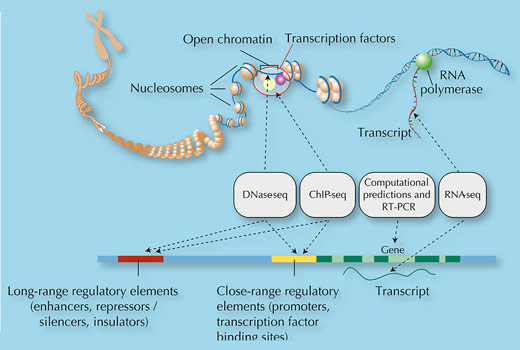
Ihre Analyse hat die Merkmale des Genoms systematisch kartiert, genauso wie eine Karte eine physische Landschaft und geographische Eigenschaften wie Wälder, Flüsse und Berge beschreibt. Die ENCODE-Wissenschaftler haben im Genom nach Merkmalen gesucht, wie Regionen, welche mit “Pssst”-Zeichen (bestimmte Arten von Methylgruppen), die eine Genstilllegung andeuten, “Bitte hier binden”-Schilder für Transkriptionsfaktoren, Antriebsregionen zur Verstärkung der Transkription, und DNA-Modifikationen, die kontrollieren, wie die DNA verpackt wird, markiert sind (Abbildung 3).
Datenflut

ENCODE-Projekts drucken
würden, würde das Papier 12
Busse füllen.
Mit freundlicher Genehmigung
von marcus_jb1973 / Flickr
Im September 2012, nach 5 Jahren voller Experimente und Analysen, die von 442 Forschern von 32 Forschungsinstituten in Großbritannien, USA, Spanien, Singapur und Japan durchgeführt wurden, hat das ENCODE-Projekt die Ergebnisse der bis dahin detailliertesten Analyse des ganzen Genoms bekanntgegeben. Die Studie hat ungefähr 300 Jahre Computerrechenzeit verschlungen, um 15 Terra-Byte an Daten (15 x 1012 Bytes) zu analysieren, die alle öffentlich zugänglich sind. Wenn die Daten mit einer Dichte von 100 Basenpaaren pro cm2 gedruckt werden würden, wäre der Papierturm 16 m hoch und länger als 30 m. Dies entspricht dem Volumen von 12 Doppeldecker-Bussen.
Das ENCODE-Projekt ist ein Beispiel dafür, was in Großprojekten erreicht werden kann, die aus den einzelnen Beiträgen von Hunderten von Wissenschaftlern zusammengebaut werden, die alle ein Teil des Puzzles hinzugefügt haben, um ein Gesamtbild des Genoms zu erhalten, was nicht durch eine einzelne Organisation möglich gewesen wäre.
Die Sequenz zum Leben erwecken
Eine der aufregendsten Erkenntnisse, die das ENCODE-Projekt gebracht hat ist, dass unser Genom voller Aktivität ist, und nicht nur eine hauptsächlich nicht-funktionelle Sequenz ist: 80% des Genoms sind aktiv. Was es genau macht, ist noch unklar, aber mit Sicherheit sind 9% (und wahrscheinlich viel mehr) an der Regulation der Genexpression beteiligt, und kontrollieren, wann und wo Proteine hergestellt werden. Die aktiven 80% des Genoms beinhalten mehr als 70 000 Promotorregionen – die „Bitte-hier-binden“-Stellen für Transkriptionsfaktoren – und fast 40 000 Verstärkerregionen – die Antriebsregionen, welche die Expression von weiter entfernten Genen kontrollieren.
Ein gewaltiges, dreidimensionales Bedienpult

eines Tontechnikers ist auch
die Kontrolle der
Genexpression kompliziert –
sie wird durch mehr als 4
Millionen Genschalter im
menschlichen Genom
kontrolliert.
Mit freundlicher Genehmigung
von Stuart Dallas Photography/
Flickr
Insgesamt hat ENCODE mehr als 4 Millionen Genschalter entdeckt, die im ganzen Genom verteilt sind. Du kannst Dir das Genom als gewaltiges Bedienpult mit vielen Schaltern, die Gene an- und ausschalten, vorstellen, wie etwa das Mischpult eines Tontechnikers. Diese Informationen vertiefen unser Verständnis der Genexpression und öffnen neue Möglichkeiten, wie man Krankheiten behandeln kann. Zum Beispiel erhöht eine kleine Veränderung in einem Genschalter, der CARD9 heißt, das Risiko an dem Crohn-Syndrom, einer entzündlichen Darmerkrankung zu erkranken, um 20%. Was wäre, wenn man Genschalter zurück zum normalen Zustand stellen könnte und damit die Ursache der Krankheit wirksam ausschalten könnte?
Die Ergebnisse von ENCODE haben auch Einblicke in die Organisation des Genoms und die physischen Interaktionen darin gewährt. Die Forscher haben herausgefunden, dass die Genschalter mit den Genen, die sie kontrollieren, in physischem Kontakt stehen, obwohl sie womöglich hunderte von Kilobasen entfernt liegen. Wir neigen dazu, uns das Genom als eine lange, gerade Reihe von Sequenzen vorzustellen, aber in Wirklichkeit liegt es dicht gepackt im Zellkern. Dabei kommen unterschiedliche Teile des Genoms in engen Kontakt miteinander.
Auf den Daten aufbauend

uns ein besseres Verständnis
der genetischen Grundlage
von Krankheiten.
Mit freundlicher Genehmigung
von AlexRaths / iStockphoto
ENCODE stellt uns eine detaillierte Karte des Genoms zur Verfügung und eröffnet neue Bereiche der Wissenschaft, die wir erforschen können. Ian Dunham vom EBI, der einer der Hauptautoren der ENCODE-Veröffentlichung ist, erklärt: „ In vielen Fällen hat man eine gute Idee, welche Gene eine Rolle in Krankheiten spielen, aber man weiß nicht genau, welche Schalter wichtig sind. Manchmal sind diese Schalter sehr überraschend – ihre Position scheint logischer für eine komplett andere Krankheit. ENCODE gibt uns eine Reihe an wertvollen Hinweisen, die wir verfolgen können, um Schlüsselmechanismen zu entdecken, die in Gesundheit und Krankheit greifen. Diese kann man ausnutzen, um komplett neue Arzneimittel herzustellen, oder vorhandene Behandlungsmethoden zu einem anderen Zweck einzusetzen.”
Die Wissenschaftler wissen jetzt nicht nur, welche Gene in einer Krankheit eine Rolle spielen, sie kennen nun auch manche der Schalter, die eine Rolle dabei spielen, wie diese Gene an- und ausgeschaltet werden. Das wird vor allem wertvoll sein, wenn es um die Interpretation von Ergebnisse von populationsbasierten Studien geht, welche Zusammenhänge zwischen Genen und Krankheiten untersuchen. Durch Verknüpfung der funktionellen Analyse von ENCODE mit Daten aus genomweiten Assoziationsstudien können Forscher die genetischen Variationen, die mit Krankheiten verbunden sind, zu den Gebieten mit regulatorischer Funktion kartieren, wie Genschalter, die durch ENCODE identifiziert wurden. Die ENCODE-Daten ermöglichen ein besseres Verständnis der genetischen Grundlage von Krankheiten und werden die Arbeit von Forschern viele Jahre lang unterstützen.
Mehr über das EBI
Das Europäisches Laboratorium für Molekularbiologie (EMBL)w1 ist eines der weltbesten Forschungsinstitute für Grundlagenforschung in Lebenswissenschaften. Das EMBL ist international, innovativ und interdisziplinär. Die Mitarbeiter aus 60 Nationen haben Hintergründe in Biologie, Physik, Chemie und Informatik, und arbeiten an Forschungsprojekten zusammen, die das ganze Spektrum der Molekularbiologie abdecken.
Das EBIw2 in der Nähe von Cambridge, UK, gehört zum EMBL. Es stellt der weltweiten wissenschaftlichen Gemeinschaft Daten aus Experimenten in Lebenswissenschaften zur Verfügung und leistet Grundlagenforschung in der Bioinformatik. Das EBI engagiert sich in der Weiterbildung von Wissenschaftlern aus Hochschulen und der Industrie, um so viel wie möglich aus der unglaublichen Datenmenge zu gewinnen, die täglich in lebenswissenschaftlichen Experimenten generiert wird.
Das EMBL ist Mitglied des EIROforumw3, dem Herausgeber von Science in School.
References
- The ENCODE Project Consortium (2007) Identification and analysis of functional elements in 1% of the human genome by the ENCODE pilot project. Nature 447: 799-816. doi: 10.1038/nature05874
-
Laden Sie den Artikel kostenfrei hier herunter, oder abonnieren Sie noch heute die Zeitschrift Nature.
-
- The ENCODE Project Consortium (2012) An integrated encyclopedia of elements in the human genome. Nature 489: 57–74. doi: 10.1038/nature11247
-
Laden Sie den Artikel kostenfrei hier herunter, oder abonnieren Sie noch heute die Zeitschrift Nature.
-
- Koutsos A, Manaia A, Willingale-Theune J (2009) Nach Genen fischen: DNA Mikroarrays in der Schule. Science in School 12.
Web References
- w1 – Erfahren Sie mehr über das EMBL.
- w2 – Erfahren Sie mehr über das EBI.
- w3 – Das EIROforum ist eine Kollaboration zwischen 8 der größten internationalen wissenschaftlichen Forschungsorganisationen Europas, die ihre Mittel, Einrichtungen und ihr Expertenwissen zusammenschließen, um die europäische Wissenschaft dabei zu unterstützen, ihr volles Potential auszuschöpfen. Als Teil seiner Bildungs- und Öffentlichkeitsarbeit veröffentlicht das EIROforum Science in School.
Resources
- Mit dem ENCODE Explorer auf der Nature-Webseite kann man in 13 Kategorien durch die ENCODE-Daten navigieren. Man kann dort auch ein free poster (20 MB) herunterladen, das einen Teil der ENCODE-Daten darstellt.
- In einem Online-Video diskutieren die Nature-Redakteurin Magdalena Skipper und Ewan Birney vom EBI über das ENCODE project.
- The Story of You: ENCODE and the Human Genome (Die Geschichte über Dich: ENCODE und das menschliche Genom) zeigt ENCODE als Cartoon.
- Ewan Birney vom EBI, Tim Hubbard vom Wellcome Trust Sanger Institut und Roderic Guigo vom CRG präsentieren ENCODE in einem video (Untertitel in Spanisch).
- Wieso probieren Sie nicht manche der folgenden Aktivitäten aus, um Bioinformatik in Ihrem Unterricht einzuführen?
-
Kozlowski C (2010) Bioinformatik mit Stift und Papier: wie man einen phylogenetischen Stammbaum aufstellt. Science in School 17.
-
Communication and Public Engagement team (2010) Can you spot a cancer mutation? Science in School 16: 39-44.
-
- Um mehr über Bioinformatik zu erfahren, siehe:
-
Hayes E (2011) An archaeologist of the genome: Svante Pääbo. Science in School 20: 6-12.
-
Pathmanathan S, Hayes E (2007) Nicky Mulder, bioinformatician. Science in School 6: 75-77.
-
Institutions
Review
Der Artikel beleuchtet eine der neuesten Fortschritte in der Humangenetik: das ENCODE-Projekt und wie die Forschungsarbeit durchgeführt wurde.
Wenn Schüler zum ersten Mal vom genetischen Code hören, sind sie zunächst einmal verblüfft, dass nur 2% der menschlichen DNA Proteine kodieren und der Rest vermutlich „Müll“ ist. Das ENCODE-Projekt hat die Funktion eines Teils dieser nicht-kodierenden DNA untersucht und herausgefunden, dass sie schließlich kein „Müll“ ist.
Wenn sie das Humangenomprojekt mit Ihren Schülern besprechen, können sie auch das ENODE-Project erklären. Es kann hilfreich sein, wenn Sie den Schülern Hintergrundinformation über Genregulation, genetische Krankheiten und ihre Behandlungen, sowie über Techniken, die in der Genetikforschung verwendet werden, zur Verfügung stellen. Der Artikel kann bei den Schülern Interesse an Bioinformatik wecken, und Sie könnten Sie ermutigen, eine Literaturstudie über das ENCODE-Projekt zu machen.
Namrata Garware, Indien





