Cómo se combate el virus de la gripe Understand article
Traducido por I. Carrero, Univ. de Valladolid. Contagiarse con el virus de la gripe puede ser algo más que una simple molestia: este patógeno ha sido capaz de producir la pandemia más mortal de la historia reciente. Claire Ainsworth nos muestra cómo trabajan los científicos para evitar que…
Imagen cortesía de EMBL Photolab
Es una batalla continua: acabas de descargarte la última actualización de un antivirus para tu ordenador y un hacker de cualquier lugar del mundo encuentra un nuevo punto débil. El ordenador está otra vez a merced de virus, spyware y demás software malicioso hasta que llegue la siguiente actualización que lo impida.
Las células de nuestro organismo se enfrentan a problemas similares: también disponen de diversos sistemas de seguridad que evitan que los virus se apropien de sus procesos bioquímicos. Pero estos sistemas no son perfectos y muchos virus despliegan estrategias casi furtivas para soslayarlos. Un grupo de científicos del proyecto de investigación FLUPOLw1 de la UE trata de dilucidar estas estrategias para poder desarrollar sistemas de seguridad para el organismo humano. Así han desenmascarado una de las tácticas que emplea el virus de la gripe y han abierto el camino a la consecución de nuevos fármacos contra futuribles pandemias de gripe.
El problema de la gripe es importante para gobiernos y organismos de salud pública de todo el mundo. Aunque las cepas víricas humanas más comunes raramente son mortales para la población general, sí que pueden ser fatales para los ancianos o para las personas que padezcan otras enfermedades o tengan debilitado su sistema inmune. Peor aún, constantemente aparecen nuevas cepas del virus que pueden llegar a ser más virulentas que las predecesoras. La amenaza más preocupante es el potencial que tiene el virus de la gripe aviar –que en principio afecta a las aves– para infectar fácilmente a los humanos. En el pasado, los virus que han podido dar este salto entre especies han sido extraordinariamente dañinos. Por ejemplo, la pandemia (del griego pan: todo + demos: pueblo; epidemia de una enfermedad infecciosa que afecta a gran parte de la población de regiones extensas, como un continente, o incluso todo el mundo) de gripe de 1918 se inició en aves y llegó a matar entre 50 y 100 millones de personas a nivel mundial. Ha sido la pandemia más mortífera de la historia reciente.
En los últimos años ha crecido el miedo en relación con la cepa H5N1 del virus de la gripe aviar (ver también Niekoop & Rienks, 2006), que ha infectado y matado a cientos de personas por todo el mundo. Si finalmente este virus adquiriera la capacidad de transmitirse de humano a humano, nos tendríamos que enfrentar a otra pandemia mortal. Existen vacunas contra la gripe, pero las que funcionan contra una cepa viral no suelen tener mucho efecto contra cepas diferentes. La producción de vacunas contra nuevas cepas lleva tiempo y se puede llegar tarde a la hora de impedir que se extienda una pandemia. Hacen falta métodos más avanzados para bloquear la propagación de los virus.
Cuando el virus de la gripe infecta una célula su finalidad es conseguir que se generen múltiples copias víricas que continúen atacando nuevas células. Una enzima del virus, la polimerasa, formada por tres subunidades, es clave en este proceso. Copia el material genético del virus y dirige la maquinaria enzimática de la célula huésped para que sintetice las proteínas virales. Esto lo hace mediante un proceso conocido como «robo de la caperuza», cap snatching, (ver imagen). La caperuza es un nucleótido modificado que se añade al principio de los RNA mensajeros (mRNA). Se requiere para dirigir la maquinaria de síntesis de proteínas celulares al punto de inicio y así poder sintetizar nuevas proteínas en función de la información contenida en el mRNA. La polimerasa viral roba la caperuza de las moléculas de RNA celular de la siguiente manera: se une a la caperuza del mRNA celular, corta esta zona y la coloca al principio del mRNA del propio virus. El modo en que la polimerasa logra esto y el papel de sus tres subunidades en el proceso son objeto de controversia. Probablemente, la bioquímica de la polimerasa viral es muy diferente a la que tienen las polimerasas humanas lo que hace que esta enzima sea un blanco muy atractivo para los fármacos antivirales puesto que esas diferencias reducen el riesgo de que la proteína humana sufra daños como efecto secundario.
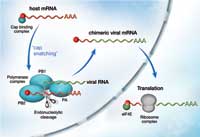
la caperuza del mRNA celular con
ayuda de la proteína PB2, que
forma parte del complejo de la
polimerasa. Con la caperuza unida,
el RNA viral puede ser traducido en
el citoplasma por los ribosomas
para dar proteínas y producir más
copias del virus. Haga clic sobre
la imagen para ampliarla
Imagen cortesía de Petra Riedinger
Por otro lado, conocer la polimerasa es crucial para comprender cómo se transmite la gripe entre especies. Varias de las mutaciones que permiten al virus de la gripe aviar infectar a humanos afectan a su polimerasa, de manera que es esencial descubrir qué efecto tienen. Esto no es fácil y requiere la participación de gran número de expertos. El proyecto FLUPOL de la UE se planteó para abordar este reto: los grupos de Stephen Cusack y Darren Hart de las dependencias del Laboratorio Europeo de Biología Molecular (EMBL, de su nombre en inglés) en Grenoblew2, Francia, junto a miembros de la Unidad sobre Interacciones Virus-Huésped (UVHCI, del nombre en inglés) de Grenoblew3 han unido sus fuerzas con equipos europeos de Madrid, Lyon, Londres y Marburgo. «Planteamos un enfoque multidisciplinar para estudiar la enzima; usamos desde la bioinformática para los aspectos estructurales hasta modelos en ratón para la transmisión interespecies», comenta Stephen.
Sin embargo, la polimerasa no revela sus secretos fácilmente. Durante años ha sido imposible obtener información detallada sobre su estructura tridimensional. Para conseguirla, los investigadores necesitan producir las proteínas que constituyen la polimerasa en su forma soluble y después cristalizarlas. Analizando con rayos X los cristales se puede deducir la estructura de esas proteínas y así comprender cómo funcionan. Para una introducción a la cristalografía, ver el artículo de Dominique Cornuéjols en este mismo número de la revista. También se puede intentar la cristalización de proteínas en clase (Blattmann & Sticher 2009).
Durante mucho tiempo nadie consiguió los cristales adecuados. Cada vez que lo intentaban, los investigadores se encontraban con que, o las proteínas formaban acúmulos insolubles e inservibles, o no se obtenía suficiente cantidad de proteína soluble para seguir trabajando. También era frustrante que los científicos no pudieran recurrir a la estrategia habitual para resolver este problema: separar las proteínas en sus componentes funcionales, denominados dominios, con la esperanza de que estos fueran solubles. Este método depende de la identificación previa de los dominios, proceso que los biólogos llevan a cabo comparando la secuencia de aminoácidos de la proteína de interés con las de proteínas con estructura conocida. Como la polimerasa del virus de la gripe es muy distinta al resto de proteínas conocidas, este abordaje se hacía imposible.

Imagen cortesía de EMBL Photolab
La solución vino con un nuevo sistema automatizado inventado por Darren. Denominado ESPRIT (Expression of Soluble Proteins by Random Incremental Truncation, Expresión de proteínas solubles por truncamiento incremental al azar), este sistema permite a los científicos investigar rápida y fácilmente todos los posibles fragmentos de los dominios solubles de interés de una proteína. En 2007, Darren y Stephen usaron ESPRIT para obtener el primer fragmento soluble de PB2, una de las tres subunidades (PA, PB1 y PB2) que forman la polimerasa del virus de la gripe (Tarendeau et al., 2007). Al determinar la estructura de este fragmento, descubrieron cómo facilita que la subunidad PB2 se incorpore al núcleo celular, donde se ensambla con las otras dos subunidades para formar la polimerasa funcional.
Stephen y Darren estaban interesados particularmente en otra región de PB2 que juega un papel clave en el robo de la caperuza. La subunidad PB2 se une al mRNA de la célula hospedadora a través de la caperuza y entonces la subunidad PB1 de la polimerasa corta la caperuza y la incorpora al principio del mRNA viral. Este mRNA modificado puede ser reconocido por la maquinaria de la célula infectada, lo que permite que se produzcan proteínas virales en vez de proteínas celulares. Sin caperuza, un mRNA no se traduce a proteínas. De esta manera, los fármacos que bloqueen la actividad que permite la unión de PB2 a la caperuza pueden detener la proliferación del virus de la gripe.
Sin embargo, hasta ahora, se sabía poco de cómo trabaja PB2. «Se había estudiado durante años, pero nadie sabía con certeza dónde estaba el dominio de unión a la caperuza», dice Stephen.
Gracias a ESPRIT, Darren, Stephen y sus colaboradores encontraron un fragmento de PB2 que se unía a la caperuza. Con ayuda de las instalaciones de cristalización de alto rendimiento del EMBL de Grenoble y de la potente emisión de rayos X del sincrotrón del Laboratorio Europeo de Radiación Sincrotrón (ESRF w4, del nombre en inglés) –ver Cornuéjols, 2009–, determinaron la estructura del dominio de unión a la caperuza de PB2 cuando la subunidad ya está unida a esa zona. Esto reveló que el dominio contiene una estructura que nunca se había visto anteriormente. «Su plegamiento es completamente diferente al de cualquier otra proteína», explica Stephen.
Pero aunque la estructura era totalmente nueva, la manera en que el dominio interaccionaba con la caperuza seguía la misma pauta que la de otras proteínas que también se unen a la caperuza: la zona de interacción es un «sándwich» que tiene dos aminoácidos de PB2 a ambos lados de la caperuza (ver imagen). «Es un bonito ejemplo de evolución convergente en el que proteínas con orígenes totalmente diferentes llegan a una misma solución», comenta Darren.
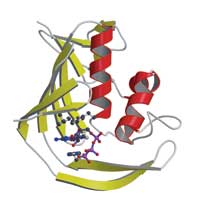
proteína PB2 del virus de la gripe
muestra cómo roba el virus la parte
de la caperuza de la célula huésped.
PB2 se une a la caperuza (marcada)
dejándola en una especie de
«sándwich» entre aminoácidos
aromáticos. Haga clic sobre la
imagen para ampliarla
Imagen cortesía de Petra Riedinger
Para probar que su fragmento era el verdadero responsable del robo de la caperuza, Darren y Stephen generaron fragmentos con mutaciones en los aminoácidos que forman el sándwich con la caperuza. Se pudo comprobar que la mayoría de los fragmentos mutantes no interaccionaba con la caperuza. Además, como demostraron los participantes en FLUPOL del Centro Nacional de Biotecnología (CNB) de Madridw5, las mismas mutaciones bloquearon la capacidad del complejo de la polimerasa para producir nuevos virus. «Fuimos capaces de probar la gran importancia del fragmento para la dinámica del virus», dice Stephen (Guilligay et al., 2008).
Ahora que se conoce la estructura, los científicos pueden basarse en ella para diseñar moléculas pequeñas y relativamente simples que bloqueen la actividad de la PB2. «Si puedes inhibir el robo de la caperuza, puedes acabar con el virus», afirma Stephen. Junto a Darren, está preparándose para buscar fármacos que puedan hacer esto. Utilizan la estructura para diseñar inhibidores pero también aprovechan la posibilidad que tienen de producir grandes cantidades del fragmento de la PB2 para buscar en amplias bases de compuestos químicos otras moléculas a las que se pueda unir esta proteína –inhibidores potenciales de PB2–. «Somos los primeros capaces de producir grandes cantidades de este fragmento y aprovechamos esta ventaja para identificar moléculas inhibidoras», explica Darren.
Recientemente, investigadores del UVHCI y del grupo de Stephen descubrieron que una parte de la subunidad PA, el tercer componente de la polimerasa viral, es responsable de la separación de la caperuza del mRNA celular (Dias et al., 2009). Este resultado fue una gran sorpresa porque todos pensaban que esa actividad residía en otra región de la polimerasa. Así, la PA ha pasado a ser un nuevo blanco para la búsqueda de fármacos antivirales.
Los investigadores produjeron cristales de ese dominio de la PA y los examinaron con la potente emisión de rayos X del ESRF de Grenoble. Las imágenes de alta resolución que obtuvieron mostraron los aminoácidos que forman parte del centro activo responsable de separar la caperuza del RNA. Un gran hueco en el centro del dominio de la PA captura las largas hebras de mRNA y un complejo metálico que hay en los bordes superiores del hueco corta la caperuza. Experimentos posteriores en el ESRF confirmaron que dicho complejo metálico contiene manganeso y este es un hecho relevante para el desarrollo de fármacos; habrá que tener en cuenta los iones metálicos a la hora de diseñar mejores inhibidores. La estrategia será aprovechar el conocimiento de la estructura de los centros activos de las subunidades de la polimerasa viral –que, o son responsables de la unión al ligando, o tienen actividad enzimática– para diseñar pequeñas moléculas con carácter inhibidor que bloqueen específicamente esos sitios y, por tanto, la reproducción del virus.
Estos hallazgos han dado a los científicos una visión más completa del mecanismo del robo de la caperuza y los investigadores están poniendo en marcha el uso de ESPRIT para desenmascarar el resto de la estructura de la polimerasa del virus de la gripe con la esperanza de que, próximamente, se abran nuevos caminos para contrarrestar las artimañas de este virus.
References
- Blattmann B, Sticher P (2009) Growing crystals from protein. Science in School 11: 30-36. www.scienceinschool.org/2009/issue11/lysozyme
- Cornuéjols D (2009) Biological crystals: at the interface between physics, chemistry and biology. Science in School 11: 70-76. www.scienceinschool.org/2009/issue11/crystallography
- Dias A, Bouvier D, Crépin T, McCarthy AA, Hart DJ, Baudin F, Cusack S, Ruigrok RW (2009) The cap-snatching endonuclease of influenza virus polymerase resides in the PA subunit. Nature 458: 914-918. doi: 10.1038/nature07745. Descargue el artículo gratuitamente aquí, o suscríbase hoy a Nature: www.nature.com/subscribe
- Guilligay D Tarendeau F, Resa-Infante P, Coloma R, Crepin T, Sehr P, Lewis J, Ruigrok RW, Ortin J, Hart DJ, Cusack S (2008) The structural basis for cap-binding by influenza virus polymerase subunit PB2. Nature Structural and Molecular Biology 15(5): 500-106. doi: 10.1038/nsmb.1421
- Niekoop L, Rienks F (2006) The ecologist’s view of bird flu. Science in School 3: 24-30. www.scienceinschool.org/2006/issue3/birdflu
- Tarendeau F, Boudet J, Guilligay D, Mas PJ, Bougault CM, Boulo S, Baudin F, Ruigrok RW, Daigle N, Ellenberg J, Cusack S, Simorre JP, Hart DJ (2007) Structure and nuclear import function of the C-terminal domain of influenza virus polymerase PB2 subunit. Nature Structural and Molecular Biology 14(3): 229-33. doi: 10.1038/nsmb1212
Web References
- w1 – Para más información sobre el proyecto FLUPOL de la UE, ver: www.flupol.eu
- w2 – Para visitar la página web de la sucursal del EMBL (European Molecular Biology Laboratory,) en Grenoble, ver: www.embl.fr
- w3 – Para saber más acerca de la UVHCI (Unit of Virus Host Cell Interactions) de Grenoble, ver: www.uvhci.fr
- w4 – Para más información sobre el ESRF (European Synchrotron Radiation Facility) de Grenoble, ver: www.esrf.eu
- w5 – Para saber más sobre el CNB (Centro Nacional de Biotecnología, Madrid), ver: www.cnb.uam.es
